导语
前几期给大家分享了微生物多样性的三个模块:OTU聚类和物种注释、Alpha多样性分析以及物种组成分析,本期主要是关于Beta多样性分析的报告解读。
Beta多样性定义为沿着环境梯度的变化物种替代的程度。不同群落或某些环境梯度上的不同点之间的共有种越少,Beta多样性就越大。作为群落结构研究的根基,常用来比较不同生态系统之间的差异,反映生物种类因环境所造成异质性。该分析包括组间距离分析、PCA分析、PCoA分析、NMDS分析、样本聚类树等。
组间距离分析
通过对不同分类或环境的多组样本的距离进行四分位计算,比较不同样本组的组内和组间的距离分布差异。同时进行multiple Student’s two-sample t-tests判断样本组间的显著性差异。进一步的将统计分析结果可视化。箱线图(Boxplot)也称箱须图(Box-whisker Plot),是利用数据中的五个统计量:最小值、第一四分位数、中位数、第三四分位数与最大值来描述数据的一种方法,它也可以粗略地看出数据是否具有对称性,分布的分散程度等信息,特别可以用于对几个样本的比较。


PCA分析
PCA(Principal Components Analysis)即主成分分析,也称主分量分析或主成分回归分析法,首先利用线性变换,将数据变换到一个新的坐标系统中;然后再利用降维的思想,使得任何数据投影的第一大方差在第一个坐标(称为第一主成分)上,第二大方差在第二个坐标(第二主成分)上。这种降维的思想首先减少数据集的维数,同时还保持数据集的对方差贡献最大的特征,最终使数据直观呈现在二维坐标系。
PCoA分析
PCoA,即主坐标分析(Principal Coordinates Analysis),可呈现研究数据相似性或差异性的可视化坐标,是一种非约束性的数据降维分析方法,可用来研究样本群落组成的相似性或相异性。采用降维思想将复杂的数据信息进行降维排序,寻找能最大程度反映样本规律的坐标系。与PCA分析基本类似,区别在于PCoA分析是基于除欧氏距离以外的其它距离。
基于UniFrac的PCOA分析也是同样的道理,Unifrac分析得到的距离矩阵可用于多种分析方法,可通过多变量统计学方法PCoA分析,直观显示不同环境样本中微生物进化上的相似性及差异性。UnifracPCoa基于进化距离,在进化水平上挖掘影响样品群落组成差异的潜在主成分。

PCA分析图
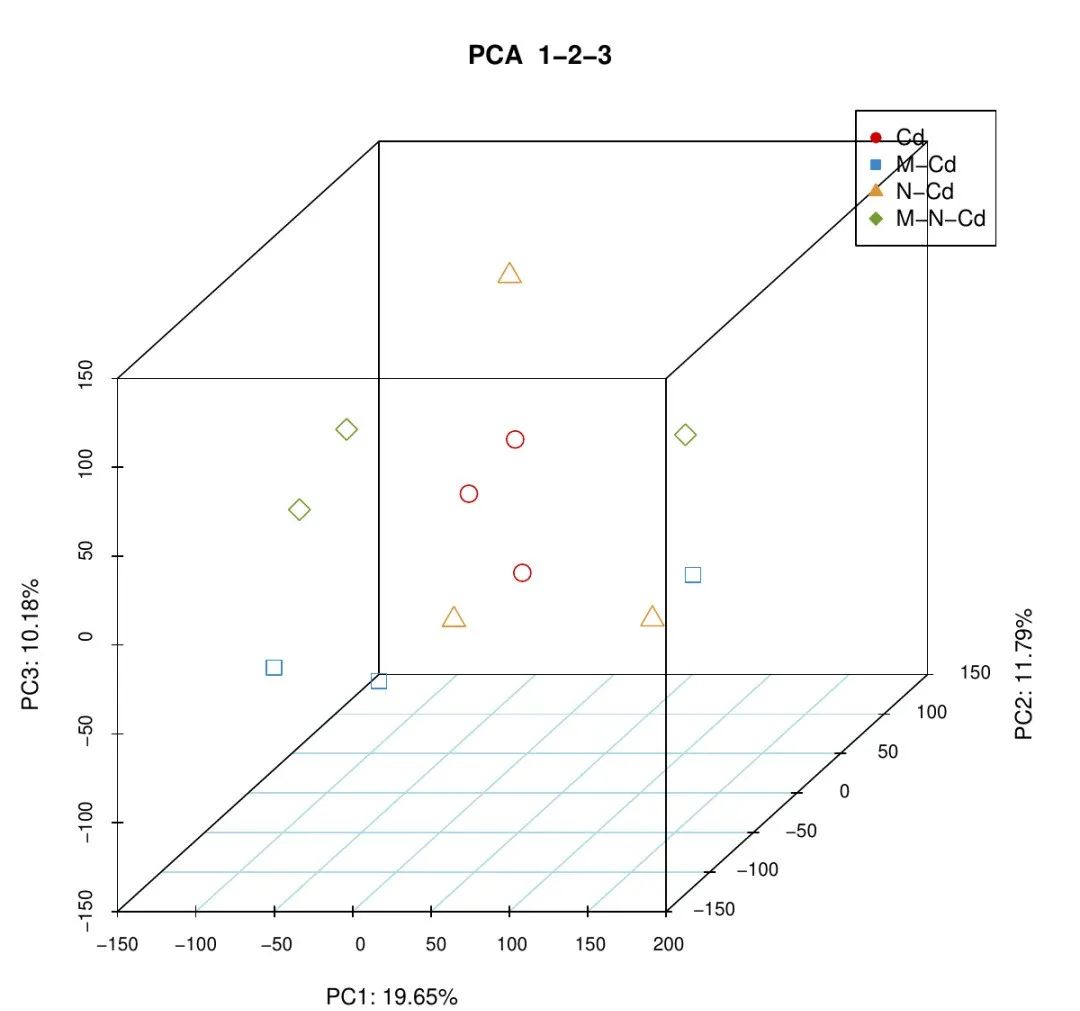
3D-PCA分析图
结果解读:横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值;纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值。图中的每个点表示一个样品,同一个组的样品使用同一种颜色表示,在PCoA分析图中,同组内的样本聚集成一簇,椭圆代表95%的置信区间,落在本组圆圈外的样本即为离群样本。如样品组成越相似,反映在PCoA 图中的距离越近。坐标轴百分比解释:如果PC1 值为50%,则表示x 轴的差异可以解释全面分析结果的50%。在元莘生物结题报告中的结果目录

NMDS分析
非度量多维尺度分析(Non-metric multidimensional scaling,NMDS)是一种将多维空间的研究对象(样本或变量)简化到低维空间进行定位、分析和归类,同时又保留对象间原始关系的数据分析方法。适用于无法获得研究对象间精确的相似性或相异性数据,仅能得到他们之间等级关系数据的情形。基于UniFrac的NMDS分析,则是反映了在进化水平上挖掘影响样品群落组成差异的程度。

