导言
微生物多样性分析中的功能预测分析是通过基于微生物组成信息,利用生物信息学工具对微生物群落的功能进行推断。这些功能预测可以帮助理解微生物在生态系统中的作用和功能,以及它们对宿主健康和环境的影响等。
本期给大家介绍的是微生物多样性之功能预测分析,主要包括KEGG功能预测和COG功能预测分析。
KEGG功能预测
KEGG(Kyoto Encyclopedia of Genes and Genomes)是一个广泛使用的生物信息学数据库,提供了关于基因组、代谢途径、生物化学反应等方面的丰富信息。KEGG功能预测的主要思想是根据微生物群落中各种微生物的相对丰度,推断这些微生物可能参与的代谢途径和功能模块。
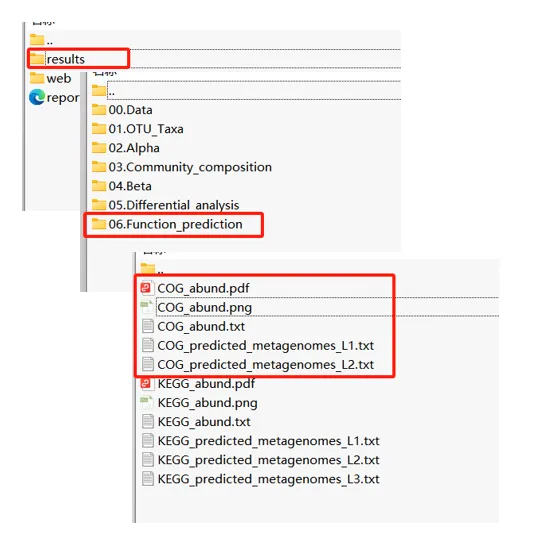
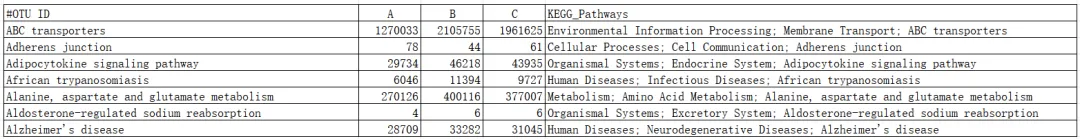
在我们的结题报告中,共提供了三个层级(level1、level2、level3)的分析结果表,以level3为例,第一列代表功能注释分类,第一行代表样本(A、B、C)以及对应的丰度信息,最后一列表示KEGG代谢通路注释信息。
通过 KEGG 代谢途径的差异分析,可以观测不同分组的样品之间微生物群落的功能基因在代谢途径上的差异和变化,是研究群落样本为适应环境变化发生的代谢功能改变的有效手段。对功能预测的结果进行可视化,下图为第二层级下 KEGG 代谢途径的差异分析图(也可以针对第三或第一层的分级进行分析)。

在元莘生物结题报告中的结果目录:
COG功能预测
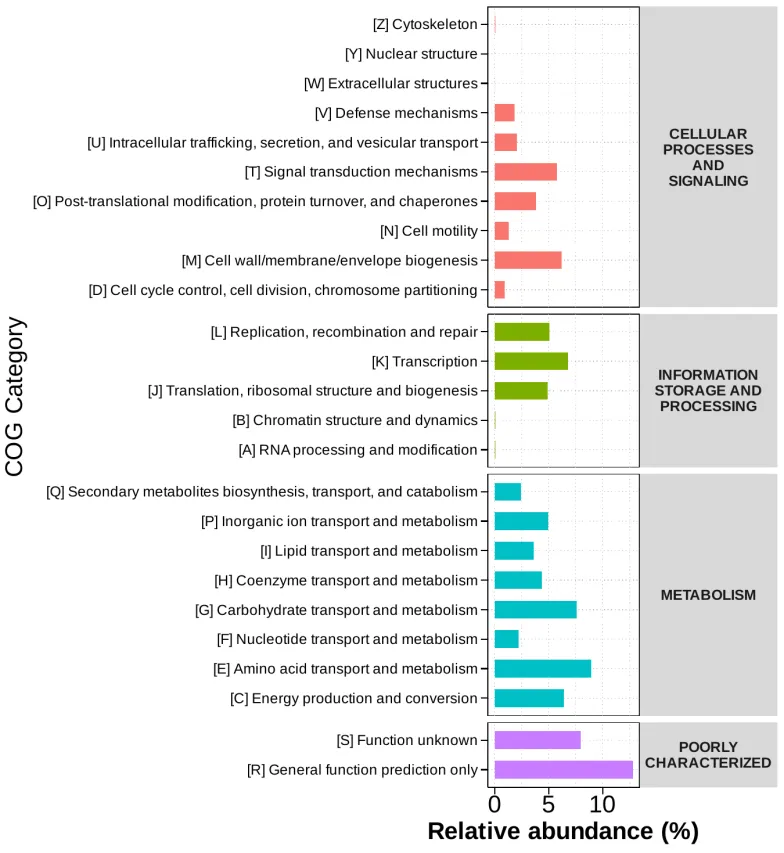
COG(Clusters of Orthologous Groups of proteins)即原核生物同源蛋白簇数据库, 是原核生物常用的蛋白功能分类数据库。COG 功能预测分析方法与 KEGG 基本相同,分析结果如下图所示,反应了样品中序列的功能分布及所占丰度。
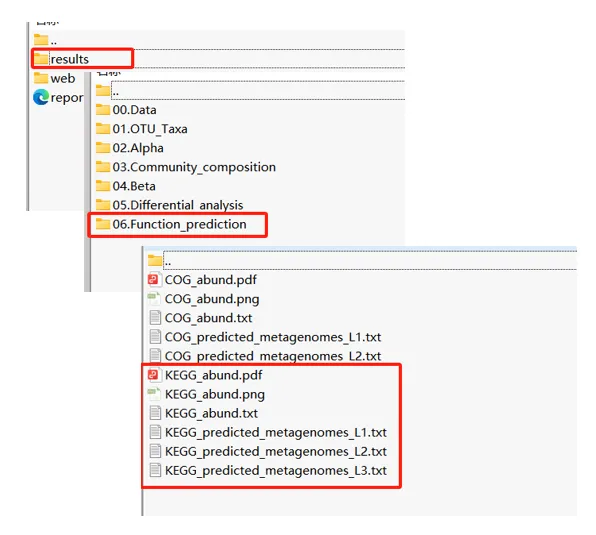
在元莘生物结题报告中的结果目录: