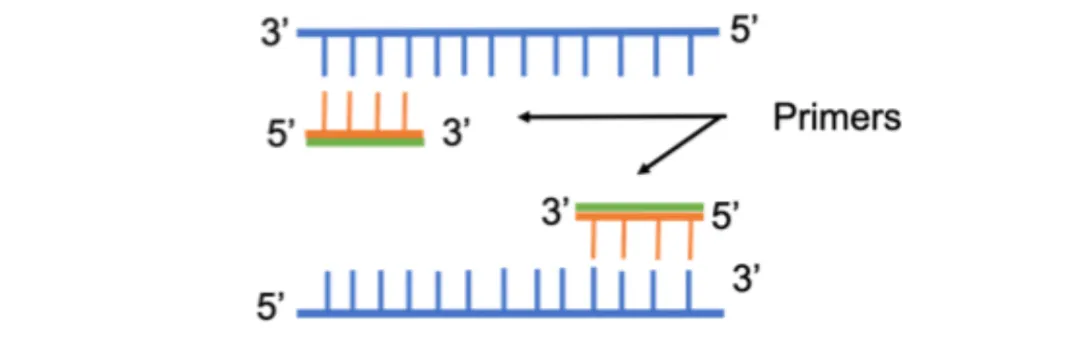
PCR引物设计是PCR实验成功的关键之一。为了帮助大家掌握这项技能,本文将详细介绍从寻找目标基因DNA到完成引物设计的全过程。
1
引物设计基本原则
引物长度:设计的引物长度应在18-25个碱基之间。过短的引物可能会导致非特异性结合,过长的引物可能会降低扩增效率。
GC含量:引物的GC含量应控制在40%-60%之间。适当的GC含量可以确保引物在PCR反应中具有适当的Tm值(熔解温度),从而保证扩增的特异性和效率。
Tm值匹配:正反向引物的Tm值应尽量接近,一般要求相差不超过2℃。Tm值过高或过低都可能导致扩增失败,最佳范围通常在50℃-65℃之间。
避免二级结构:引物设计时应避免形成发夹结构、二聚体或其他二级结构,这些结构可能干扰PCR反应。
2
寻找目标基因DNA序列
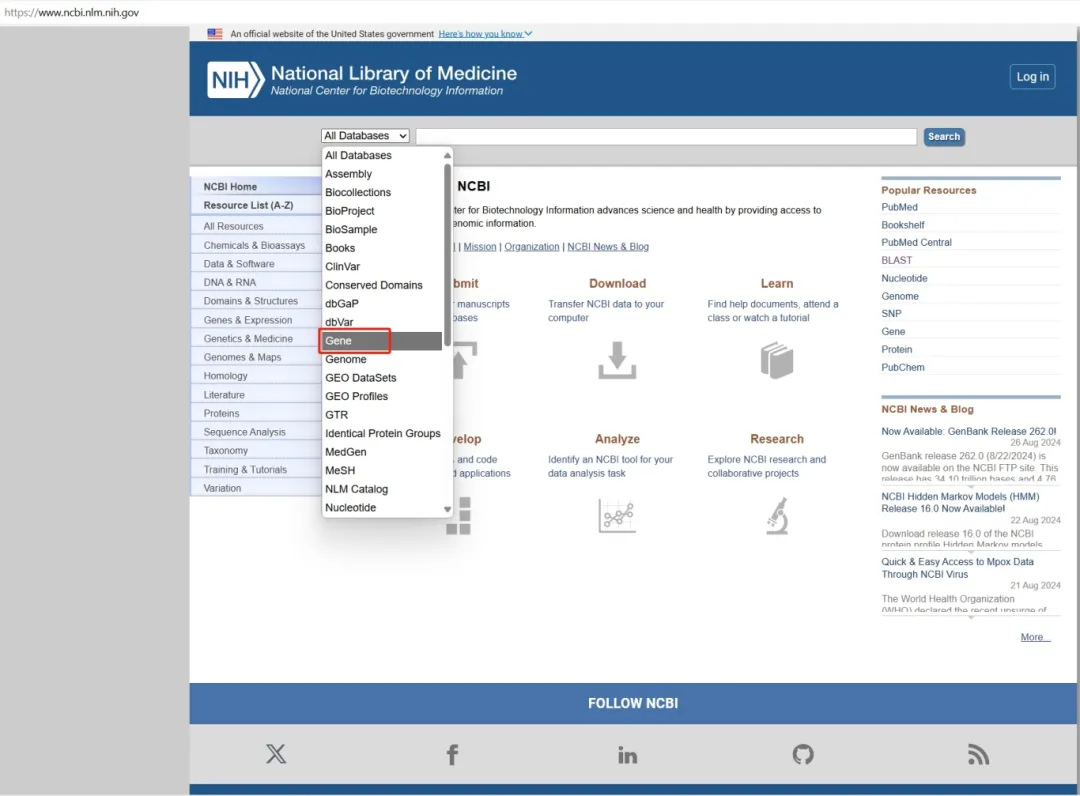
步骤1:访问NCBI数据库
打开浏览器,进入NCBI网站(https://www.ncbi.nlm.nih.gov/)。
在页面顶部的搜索框中,输入目标基因的名称,并选择“Gene”数据库。
步骤2:选择目标基因序列
在搜索结果中查找并选择目标基因的种属。确保选择的是目的种属的基因以及序列,以便设计引物时能够覆盖基因的关键区域。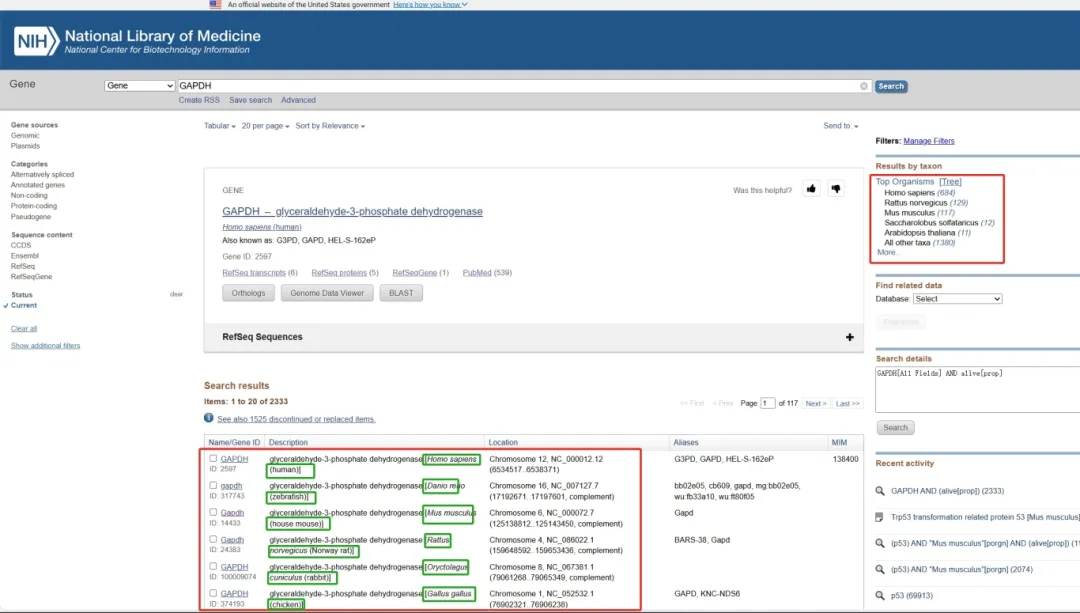
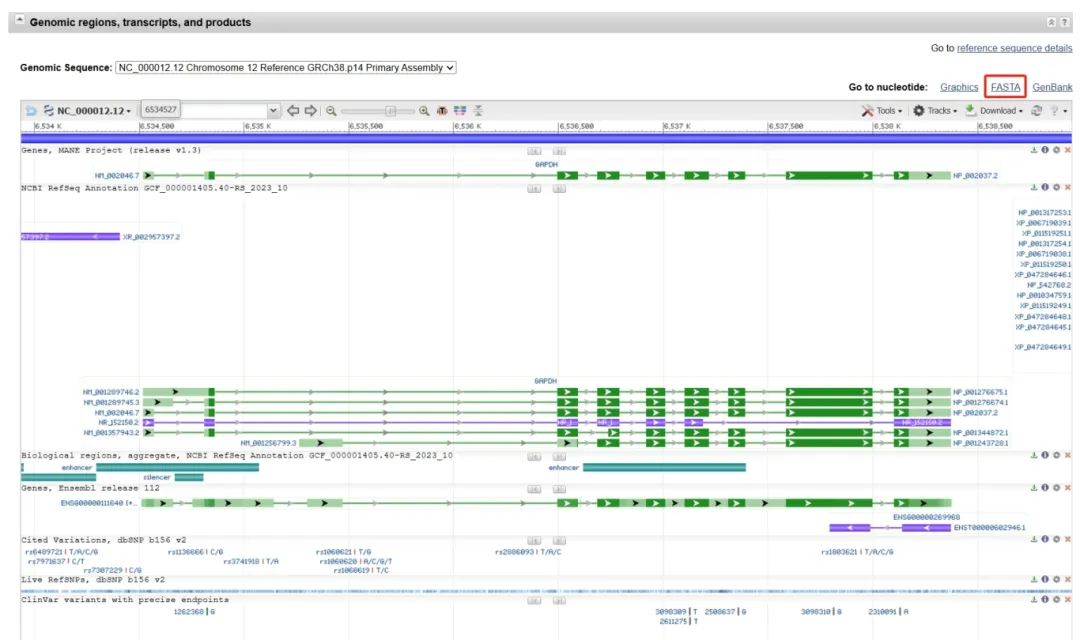
步骤3:获取DNA序列
点击进入该基因的详细信息页面。
在页面中找到FASTA格式的序列部分,通常在页面下方“Genomic regions, transcripts, and products”选项卡中。
复制这段完整的DNA序列(只包括碱基,不包括行号和其他字符)。
3
使用Primer3设计基因的引物
步骤1:访问Primer3 Plus
打开浏览器,进入Primer3 Plus网站(http://primer3.ut.ee/)。
这个网站提供了一个简单而强大的引物设计界面,适合大多数PCR引物的设计需求。
步骤2:输入目标DNA序列
在Primer3 Plus的主页上,你会看到一个大的文本框,标注为“Paste source sequence below”或类似描述。将之前从NCBI复制的目标基因序列粘贴到这个文本框中。
并且在Select species中填写正确的物种。
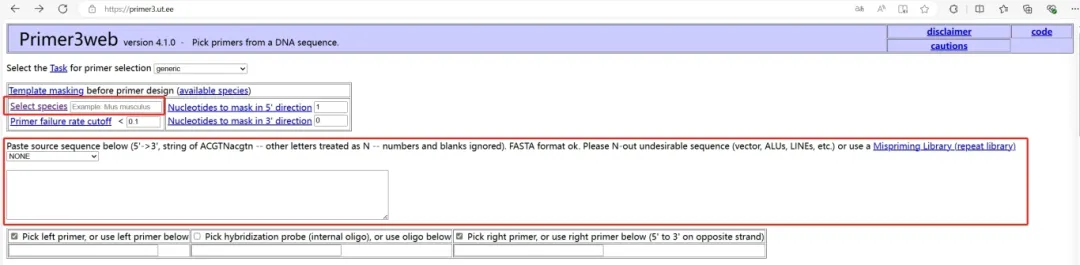
步骤3:设定设计参数
在页面下方,你会看到多个可以调整的参数选项:
Primer size:设置引物的长度范围为18-25个碱基(默认即可)。
Primer Tm:设置Tm值范围为50℃-65℃。这有助于选择最佳的引物对。
Primer GC%:设置GC含量为40%-60%(默认即可)。
Product size range:输入你希望扩增的DNA片段大小范围(例如100-200 bp)。
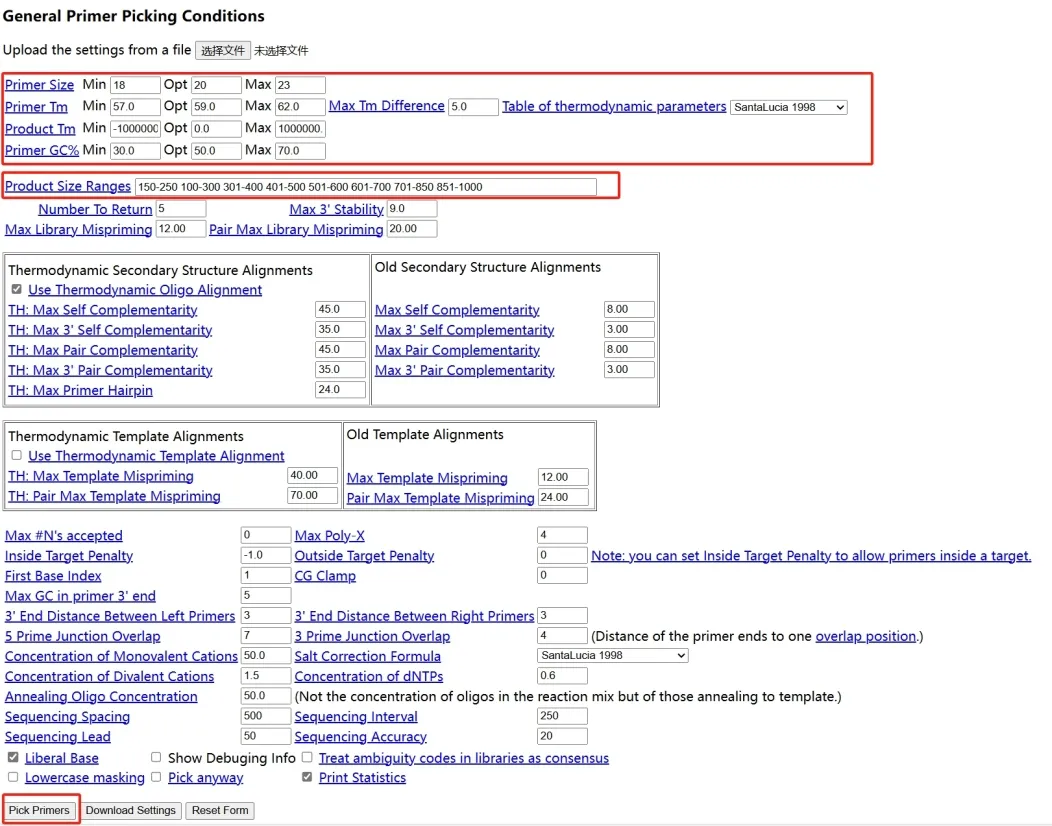
步骤4:生成引物
确认所有参数设置无误后,点击页面底部的“Pick Primers”按钮。
Primer3将根据你输入的参数,自动生成几对适合的引物。
步骤5:评估和选择引物
在生成的引物列表中,你会看到每对引物的详细信息,包括Tm值、GC含量、引物序列、扩增产物大小等。
选择一对Tm值接近、GC含量合适、且不容易形成二级结构的引物作为最终的PCR引物。
04
检查引物的特异性
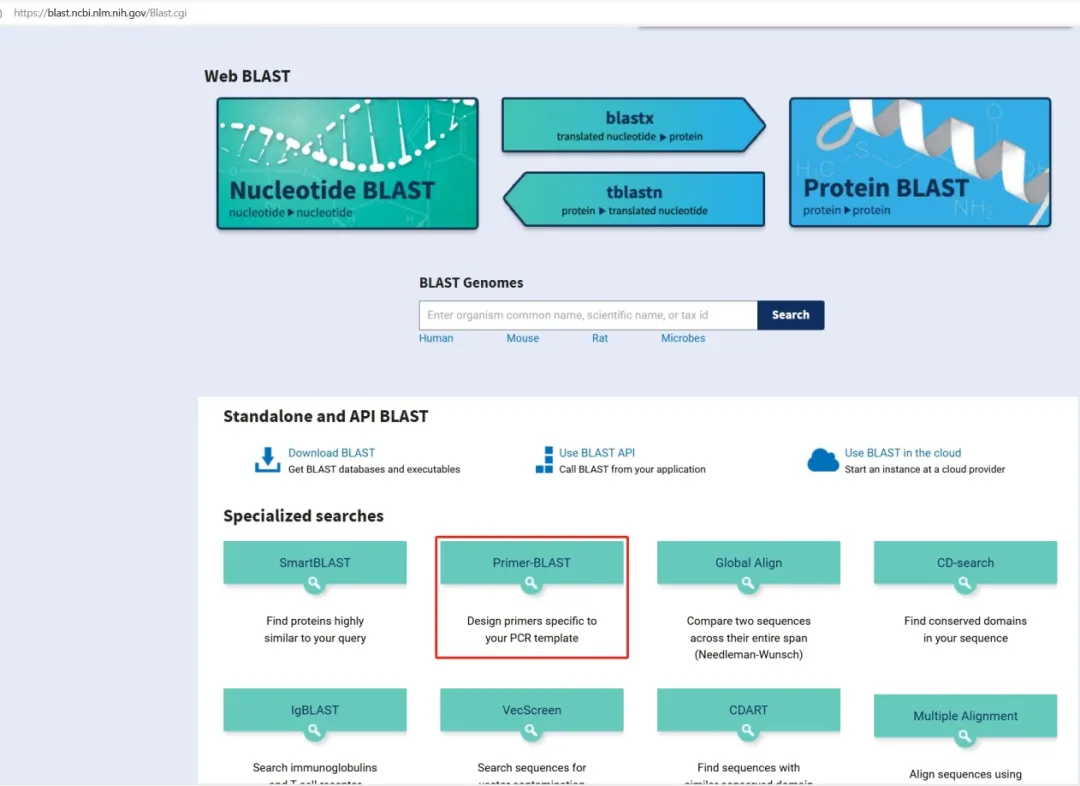
步骤1:使用NCBI的BLAST工具
打开浏览器,进入NCBI BLAST网站(https://blast.ncbi.nlm.nih.gov/Blast.cgi)
在BLAST主页上,选择“Primer-BLAST”工具,这是一个专门用于检测引物特异性的工具。
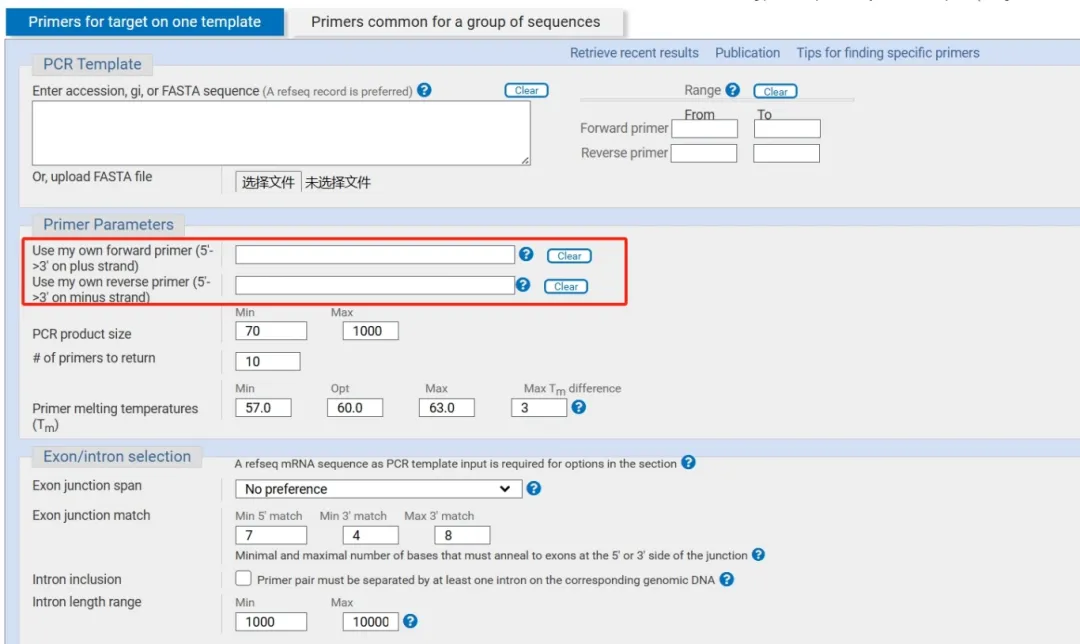
步骤2:输入引物序列
在Primer-BLAST的页面中,分别将你设计的正向引物和反向引物序列粘贴到对应的输入框中。
在Organism选择你研究的物种,以确保引物只与目标基因相匹配。
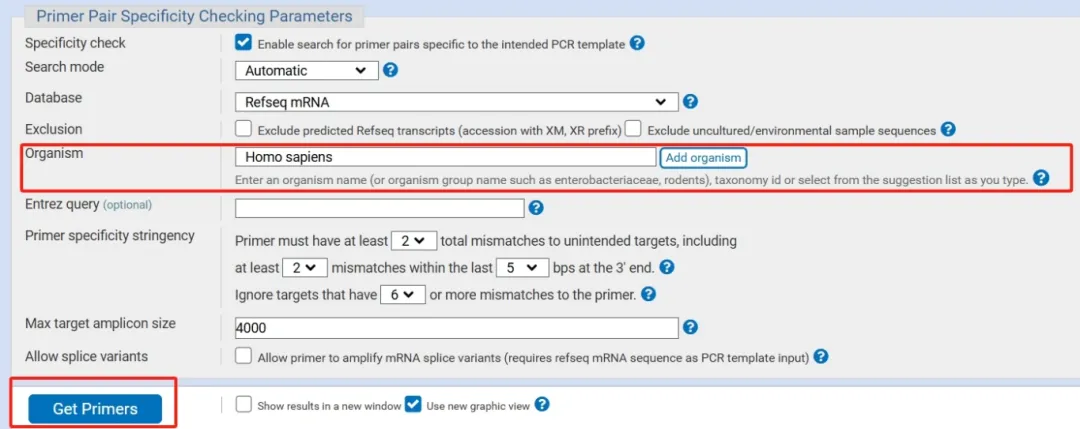
步骤3:运行BLAST分析
点击“Get Primers”按钮,运行引物特异性分析。这个过程可能需要几秒钟到几分钟,取决于数据库的繁忙程度。
步骤4:检查结果
在结果页面中,查看引物的匹配情况。确保引物只与目标基因序列有特异性结合,而不会与其他基因发生非特异性匹配。
05
引物设计的注意事项
3'末端匹配:引物的3'末端必须严格避免与非目标序列的意外配对,因为3'末端是DNA聚合酶延伸的起点,任何非特异性结合都可能导致错误的扩增。
避免连续重复序列:设计引物时要避免在引物序列中出现连续的G或C,例如“GGGG”或“CCCC”,因为这些重复序列容易引发引物的非特异性结合。
二级结构预测:在使用引物之前,使用OligoAnalyzer等工具检查引物是否可能形成发夹结构或二聚体。如果检测到有这些结构,应重新设计引物。
本期就到这里啦,敬请期待下一期!
