导语
DNA从线粒体逃逸到核基因组 (核线粒体DNA, NUMT) 是一个持续的过程。虽然在真核生物基因组中普遍观察到,但它们在哺乳动物范围内的进化轨迹却知之甚少。由于NUMT在过去2亿年间的快速进化和染色体重排,主要的挑战在于跨物种的同源性分配。近日,《Molecular Biology and Evolution》在线发表了一篇题为“Comparative Genome Microsynteny Illuminates the Fast Evolution of Nuclear Mitochondrial Segments (NUMTs) in Mammals”的文章。该文章系统研究了45种哺乳动物基因组中NUMT插入的特征,并建立了一种新的基于同质性的方法来准确预测同源NUMTs,并确定它们在哺乳动物中的进化。让我们一起来学习一下吧~

文献标题:Comparative Genome Microsynteny Illuminates the Fast Evolution of Nuclear Mitochondrial Segments (NUMTs) in Mammals
发表期刊:Molecular Biology and Evolution
影响因子:10.7
发表时间:2024.01.03
研究思路

研究结果
1. 哺乳动物基因组中的 NUMT 插入概述
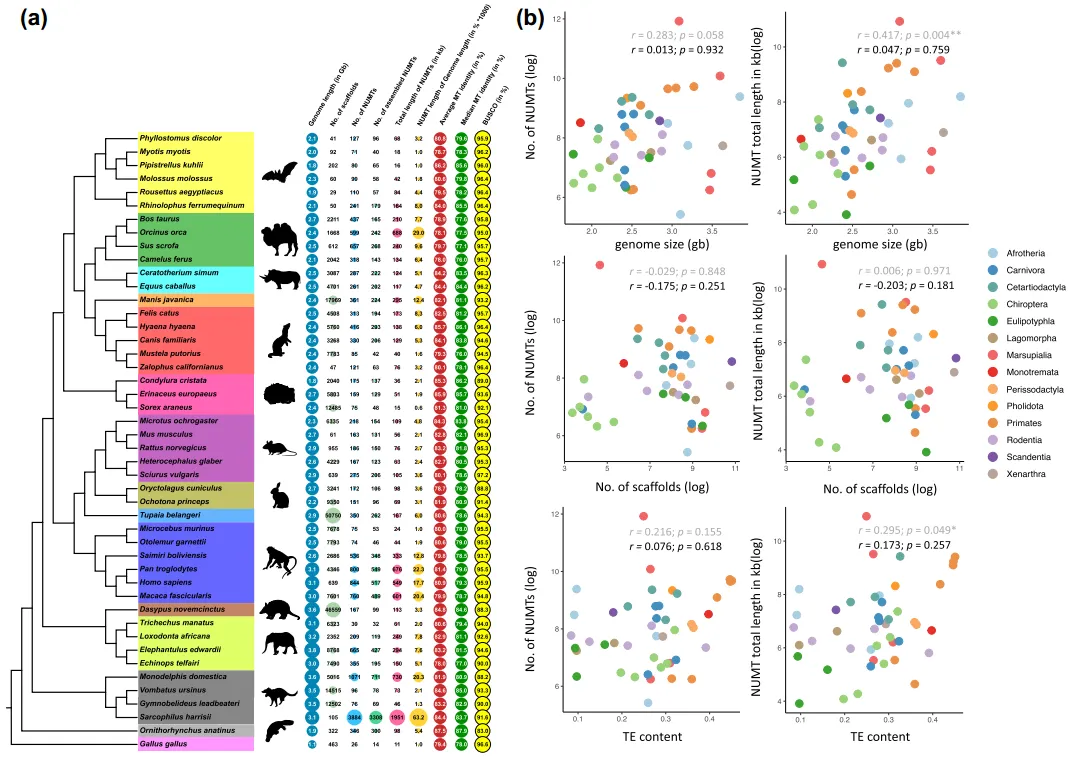
哺乳动物基因组中NUMT 插入的数量从 43 个(海牛Trichechus manatus)到 3,886 个(袋獾Sarcophilus harrisii),中位数为 218,与之前研究报告的数量相当。尽管不同物种的 NUMT 数量差异很大,但平均只占基因组的不到 0.01%。袋獾(S. harrisii)基因组中的累积 NUMT 长度最长(1,951 kb),而普通鼩鼱(Sorex araneus)的基因组中的 NUMT 长度最短(15.07 kb)。不同物种的个体 NUMT 长度从 30 bp 到 16,699 bp 不等,在同一目物种中的分布情况也类似。在 45 个基因组中,有 20 个基因组拥有超长的 NUMTs(超过 10 kb),其中一些几乎来自整个 mtDNA。有趣的是,鲸偶蹄目Cetartiodactyla 中的物种具有最高比例的复杂 NUMT 块,这些块由位于 2 kb 基因组距离内的多个单个 NUMT 组成。为了证明预测NUMT 片段的可靠性,以Molossus Molossus基因组为例,其最大NUMT片段的基因组位点得到了PacBio原始读数的支持。作者注意到,NUMT累积长度分别与基因组大小(P = 0.004)和基因组转座元件(TE)含量(P = 0.049;特别是长穿插核元件[LINEs],P = 0.033)呈正相关。然而,跨物种相关性检验应考虑进化关系,在对这45 种哺乳动物的系统发生进行校正后,检验的显著性下降了(P = 0.759 和 P = 0.257)。除了在近缘物种中观察到相似的NUMT长度分布外,这些结果显示NUMT插入在一定程度上表明了哺乳动物的系统发育,并可能成为系统发育推断的潜在遗传标记。
2. 灵长类和有袋类物种的 NUMT 扩展
在灵长动物和有袋动物中观察到了 NUMT 插入的爆发。在灵长目中,有 4 个物种的 NUMT 数量较多(547 至 846 个),从而形成了类人猿亚目 Haplorhini,而在姊妹原猴亚目 Strepsirrhini 中研究的另外 2 个物种只有少量的 NUMT 插入(76~77 个)。这表明,在Haplorhini与Strepsirrhini分化之后,NUMT插入发生了爆发。有袋目动物和袋獾(S. harrisii)的观察结果类似,最近的一项研究发现袋鼬科 Dasyuridae中的两个物种,即袋獾(S. harrisii)和黄足蜈蚣(Antechinus flavipes),也有快速的 NUMT 扩张。然而,负鼠科(Didelphidae)的南方短尾负鼠(Monodelphis domestica)经历了与袋獾类似的NUMTs爆发(1,083个),这与负鼠科(Petauridae)和袋獾科(Vombatidae)的利氏袋鼯(Gymnobelideus leadbeateri)(76个)和塔斯马尼亚袋熊(Vombatus ursinus)(112个)相反。这一结果表明,在有袋类动物的进化过程中,NUMT含量的大规模扩张可能发生过多次,而彻底的分类对于得出NUMT扩张的准确结论至关重要。
3. 产生NUMT的mtDNA区域是非随机的
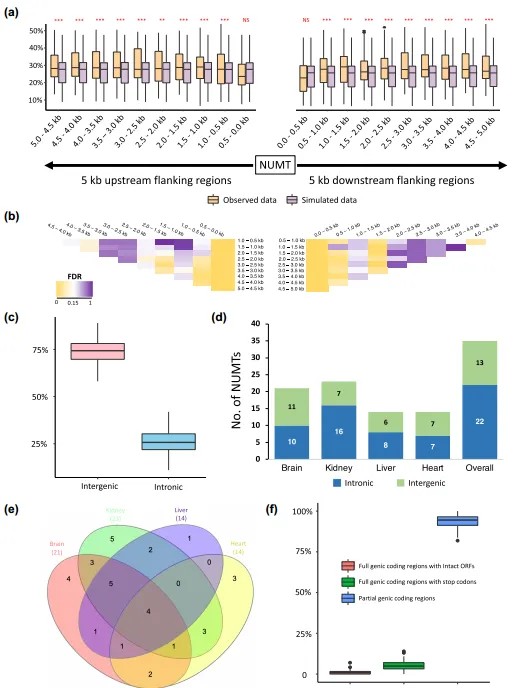
据报道,在少数灵长类动物基因组中,mtDNA的某些区域,如D-loop区,往往不会产生NUMTs,而在牛和少数鲸目动物基因组中,NUMTs却能很好地代表这些区域。通过以每个物种 50 bp 的滑动窗口扫描线粒体基因组(320 个窗口代表每个物种 16,000 bp的mtDNA),使用 Mann-Whitney U检验比较了 45 个物种中所有可能窗口的NUMTs mtDNA覆盖率。结果显示,NUMT 覆盖率在不同窗口之间的分布各不相同。在51,040次比较中,9273 次(18.17%)测试得出了显著结果,一些窗口(如w1至w50对应mtDNA 上的第1至2500个bp;w200至277对应 mtDNA 上的第10,000至13,850 个bp)的覆盖率分布与其他窗口不同。为了进一步验证这些结果,对每个物种的 NUMTs的mtDNA坐标进行了随机重新排列,从而模拟了等覆盖率零分布的覆盖率数据,在各窗口之间进行了配对比较,并重复这些分析1000次。结果显示,从观测数据中得出的显著检验次数(9273次)是模拟数据的10倍。这一结果支持了NUMTs 来源的mtDNA 区域是非随机的这一假设。此外,在单核苷酸分辨率下,NUMTs的mtDNA覆盖范围在不同类群中呈现出物种特异性模式。

4. NUMT可能存在于转座子富集区和基因间区,大多数没有表达,也没有功能
先前的证据表明,NUMT似乎插入到TE含量丰富的基因组位置。为了进一步研究哺乳动物之间的这种关联,首先调查了每个 NUMT/NUMT 区块 5 kb 侧翼区域(上游和下游)的 TE 含量,窗口大小为 500 bp。虽然各目之间的分布不同,但基于窗口的分析显示,NUMT 的 5 kb 侧翼区域的 TE 含量在不同物种之间呈现出一种独特的模式。除了第一个上游和下游窗口外,所有窗口的 TE 含量都明显高于背景平均 TE 含量(P < 0.05)(Mann-Whitney U 检验)。在不同物种中,这两个与 NUMTs 直接相连的窗口的 TE 含量明显低于两端长达 5 kb 的其余侧翼窗口(P < 0.05,Mann-Whitney U 检验)。接下来,研究了NUMT是否容易位于内含子或基因间区域。在有高质量基因组注释的 36 种哺乳动物中,在基因间区检测到的 NUMTs 数量明显多于内含子区,比例为 3 比 1(P < 0.001)。苍白矛鼻蝠(Phyllostomus discolor)基因间 NUMTs 的比例最高(89%),而鸭嘴兽(Ornithorhynchus anatinus)基因内 NUMTs 的比例最高(41.9%)。据估计,动物基因组内含子和基因间区的累积大小相似,但该研究的结果表明,NUMTs往往位于基因间区。
一旦整合到核基因组中,NUMTs通常被认为是无功能的。为了证实这一假设,检测了 5 个物种的 4 种组织类型中的 NUMT 表达。结果显示,NUMTs很少表达,平均只有一小部分(0.15% 至 4.51%)在这些物种的不同组织中表达。以鸭嘴兽为例,观察到表达的 NUMTs 显著富集在内含子中。推测内含子NUMTs可以在没有独立启动子的情况下与其宿主基因共表达。这一假设也可以解释为什么鸭嘴兽中表达的 NUMTs 比例很高,因为其基因组中内含 NUMTs 的比例最高。值得注意的是,鸭嘴兽中具有个体特异性的多态 NUMTs,这在哺乳动物基因组中很常见。此外,有 4 个 NUMTs 在鸭嘴兽的所有 4 种组织类型中都有表达,这表明它们受到了一些选择性限制。需要注意的是,由于不同个体的 NUMT 存在多态性,因此低估了不同物种中表达 NUMT 的百分比。因此,由于每个物种的基因组测序和 RNA-Seq 的样本来源不同,检测个体特异性 NUMTs 的表达具有挑战性。然而,结果显示,根据核遗传密码,不同物种中平均 99.2% 的 NUMTs 包含不完整的 mtDNA 基因开放阅读框(ORF)或带有终止密码子的完整基因编码区。尽管这些基因可以表达,但它们不太可能被翻译成蛋白质或其他物质。
研究总结
该文章系统地研究了 45 个哺乳动物基因组中 NUMT 插入的特征,并建立了一种新颖的、基于同源关系的方法来准确预测同源 NUMT 并确定它们在哺乳动物中的进化。通过一系列跨类群的比较分析,发现了NUMTs可能来自mtDNA中的非随机区域,很可能存在于转座子富集区和基因间区,而且不太可能编码功能蛋白。利用基于同源关系的方法,该研究对630对全基因组微共线关系进行了比较,预测了36种哺乳动物的NUMTs同源关系。根据 NUMTs 在不同类群中存在与缺失的系统发育模式,利用聚合方法构建了哺乳动物中 NUMTs 的祖先状态。结果显示,劳亚兽总目Laurasiatheria 中发现了猛兽真有蹄类群Fereuungulata的祖先节点,而其隶属关系仍存在争议。这项研究拓宽了我们对哺乳动物基因组中 NUMT 插入和进化的认识,并突出了 NUMTs 作为系统发育推断中替代遗传标记的优点。
参考文献
Uvizl M, Puechmaille SJ, Power S, Pippel M, Carthy S, Haerty W, Myers EW, Teeling EC, Huang Z. Comparative Genome Microsynteny Illuminates the Fast Evolution of Nuclear Mitochondrial Segments (NUMTs) in Mammals. Mol Biol Evol. 2024 Jan 3;41(1):msad278. doi: 10.1093/molbev/msad278. PMID: 38124445; PMCID: PMC10764098.