小伙伴们大家好,上期内容我们详细了解了非靶代谢组研究中重要的质控环节。
今天,让我们来看看非靶代谢组报告中代谢物鉴定和组间差异分析的部分吧~
一、鉴定结果统计与分析
将项目鉴定到的所有代谢物(合并正负离子鉴定到的代谢物)根据其HMDB数据库的化学分类(Chemical Taxonomy)归属信息进行分类统计,各类代谢物数量所占比例如图1所示。

图1 鉴定的代谢物在各化学分类的数量占比
二、组间差异分析单变量统计分析是从某单一变量水平考察组内变异度和组间差异,而多维统计分析是从总体水平反映组间差异以及反映组内的变异度。代谢组数据具有高维度且变量间高度相关的特点,运用传统的单变量分析无法快速准确地挖掘数据内潜在的信息,所以需要运用多元统计的方法,如PCA、OPLS-DA分析等分析,在最大程度保留原始信息的基础上对采集的多维数据进行降维分析。
同时结合单变量统计分析和多维统计分析结果,是代谢组最常使用的一种组间显著性差异代谢物筛选方法。这些显著性差异代谢物可能是潜在的生物标志物。通过研究差异代谢物参与的代谢通路,逆推找出调节酶和基因,有助于揭示其参与的生命活动机制,完成调控通路等方面的研究。
1) 单变量统计分析
单变量统计分析方法是最常用的统计分析方法之一。在进行两组样本间的差异分析时,常用的单变量统计分析方法包括变异倍数分析(Fold Change Analysis,FC Analysis)、T检验/非参检验。
基于单变量分析,对检测到的所有代谢物(含未被鉴定的代谢物)进行差异分析。将符合FC>1.5或FC<0.67的代谢物作为差异代谢物,采用火山图的形式来进行可视化展示,结果如图2所示。
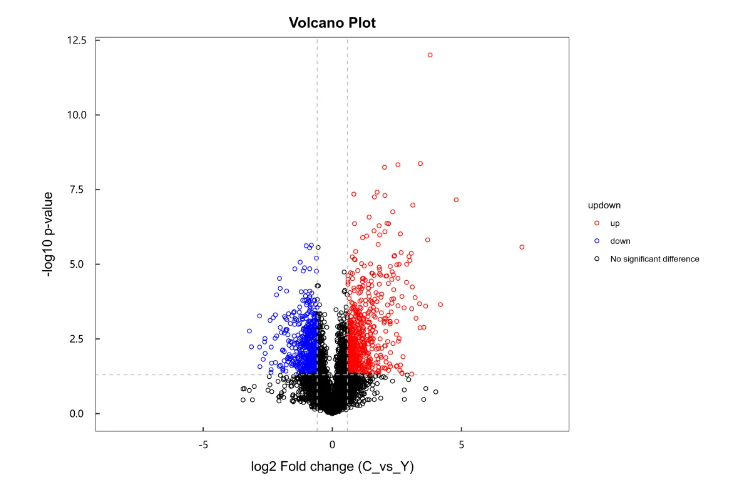
图2 火山图
注:图中横坐标为差异表达倍数(Fold Change)的log2的对数值,纵坐标为显著性 p value 的 -log10 的对数值。显著差异代谢物:满足FC>1.5、p value < 0.05 代谢物用红色表示,满足FC<0.67、p value <0.05的代谢物用蓝色来表示,非显著差异代谢物用黑色来表示。
为了直观便于观察差异代谢物的分类归属,可以使用不同颜色来做区分,结果如图 3 所示。

图3 火山图(颜色与差异代谢物化学分类相关)
注:图中横坐标为差异表达倍数(Fold Change)的log2的对数值,纵坐标为显著性 p value的-log10的对数值。有定性的差异代谢物按照其化学分类归属信息,用不同的颜色来区分。没有化学分类归属的差异代谢物定义为 undefined,非显著差异代谢物用黑色来表示。
2)多维统计分析
① 主成分分析(Principal Component Analysis, PCA)
PCA是一种无监督的数据分析方法,通过重新组合原始代谢物数据,形成新的综合变量集合。这些新变量被选取为能够最大程度上反映原始数据的信息,从而实现数据降维。应用PCA对代谢物数据进行分析,不仅能够捕捉样本之间和样本内的变异情况,还能帮助揭示样本之间整体分布趋势及其差异。因此,在数据分析中,通常首先使用PCA方法,以探索和理解样本数据的整体结构和组间样本的差异情况。
分别对各个比较组做PCA分析,以示例对比组为例,PCA得分图见图 4。
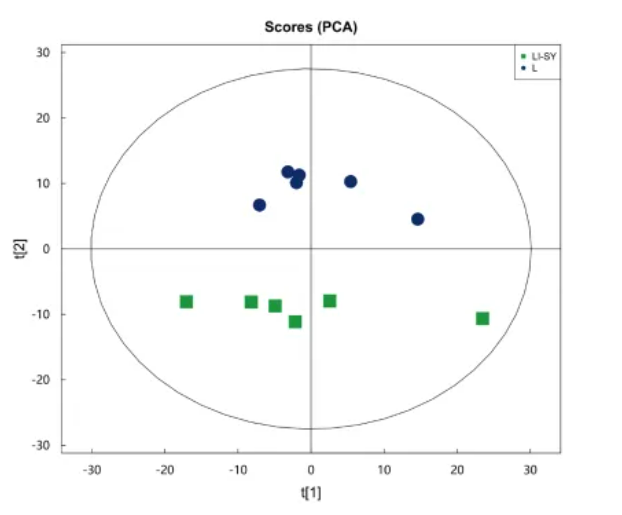
图4 PCA 得分图
注:图中横坐标 PC [1] 代表主成分1,纵坐标 PC [2] 代表主成分 2,椭圆代表 95%置信区间。同一颜色的点表示组内的各个生物学重复,点的分布状态反映出的是组间、组内的差异度。
组内聚集度高,组间分离明显,说明模型可靠,组间有明显差异。
②偏最小二乘判别分析(Partial Least Squares Discrimination Analysis, PLS-DA)
PLS-DA是一种有监督的判别分析统计方法,该方法运用偏最小二乘回归建立代谢物表达量与样品类别之间的关系模型,来实现对样品类别的预测;其与 PCA 原理类似,但在分析时必须对样品进行指定分组,这种模型计算的方法强行把各组分门别类,有利于发现不同组间的异同点。对于组间差异不够明显的样品,采用PCA方法常常无法区分样品的组间差异,这种情况下采用PLS-DA模型可能更加有效。
示例对比组PLS-DA得分图:
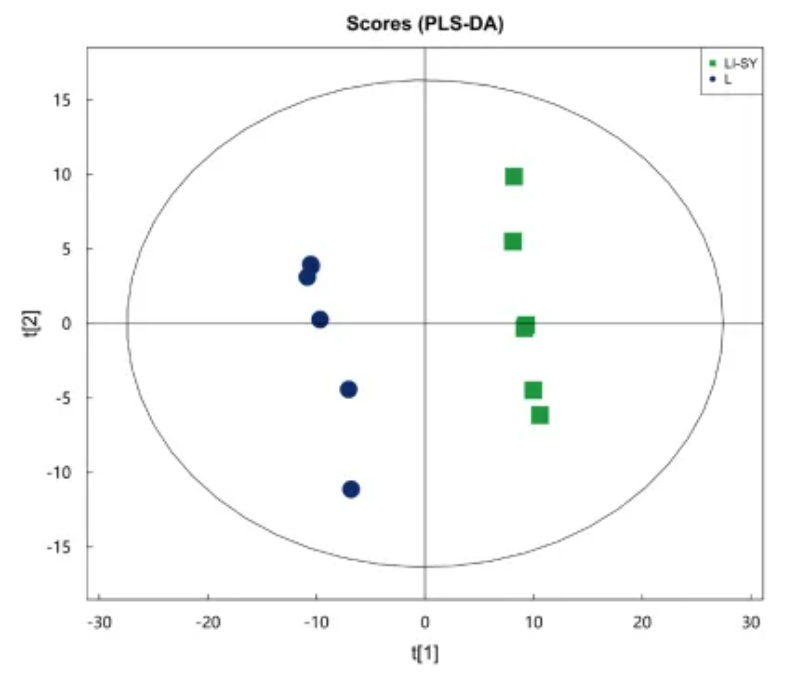
图5 PLS-DA 得分图
为避免有监督模型在建模过程中发生过拟合,采用置换检验(Permutation test)对模型进行检验,以保证模型的有效性。图6显示了示例对比组OPLS-DA 模型的置换检验图,随着置换保留度逐渐降低,随机模型的R2和Q2均逐渐下降,说明原模型不存在过拟合现象,模型稳健性良好。
示例对比组PLS-DA置换检验:
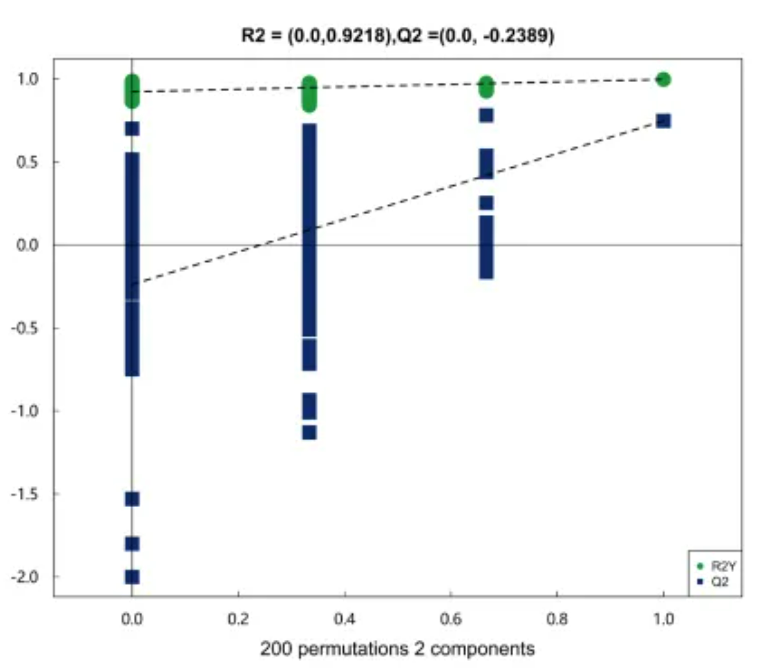
图6 PLS-DA 置换检验图
注:模型质量评估标准:左侧的所有蓝色Q2值都低于右侧的原始点,或者Q2点的蓝色回归线与垂直轴 (左侧)在零或以下相交。
③ 正交偏最小二乘判别分析(Orthogonal Partial Least Squares Discriminant Analysis, OPLS-DA)
OPLS-DA是一种对PLS-DA进行修正的分析方法,可以滤除与分类信息无关的噪音,提高了模型的解析能力和有效性;在OPLS-DA得分图上,有两种主成分,即预测主成分和正交主成分。OPLS-DA将组间差异最大化的反映在t[1]上,所以从 t[1] 上能直接区分组间变异,而在正交主成分to[1]上则反映了组内的变异。示例对比组的OPLS-DA模型得分图见图7,可见OPLS-DA模型能区分两组样本。
示例对比组OPLS-DA得分图:
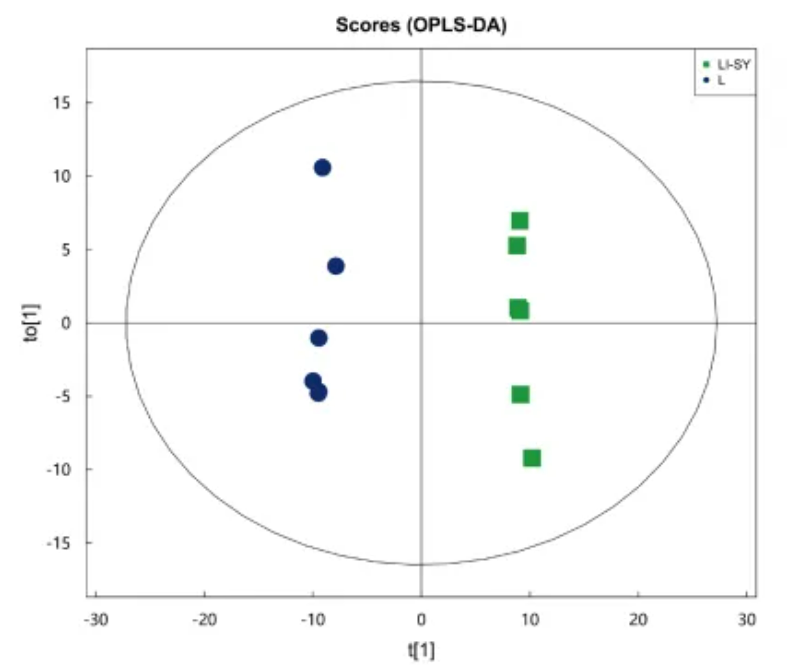
图7 OPLS-DA 得分图
注:图中 t[1]代表主成分1,to[1]代表主成分2,椭圆代表95%置信区间。同一颜色的点表示组内的各个生物学重复,点的分布状态反映出的是组间、组内的差异度。
为避免有监督模型在建模过程中发生过拟合,采用置换检验(Permutation test)对模型进行检验,以保证模型的有效性。图8显示了示例对比组 OPLS-DA 模型的置换检验图,随着置换保留度逐渐降低,随机模型的R2和Q2均逐渐下降,说明原模型不存在过拟合现象,模型稳健性良好。
示例对比组OPLS-DA置换检验:

图8 OPLS-DA 置换检验图
注:模型质量评估标准:左侧的所有蓝色 Q2 值都低于右侧的原始点,或者 Q2 点的蓝色回归线与垂直轴 (左侧)在零或以下相交。
三.筛选显著性差异代谢物
书接上文,OPLS-DA 模型除能够对多维数据进行降维分析外,其得到的变量权重值(Variable Importance for the Projection, VIP)还能够用于衡量各代谢物的表达模式对各组样本分类判别的影响强度和解释能力,有助于挖掘具有生物学意义的差异代谢物分子。通常,VIP>1的代谢物被认为在模型解释中具有显著贡献。
代谢组学通常以严格的 OPLS-DA VIP>1和T检验p value <0.05为显著性差异代谢物筛选标准。表中,用黄色背景色表示的是差异代谢物,若筛选到的差异代谢物数量较少,可考虑将标准降低,调整为OPLS-DA VIP>1 和p value < 0.1。
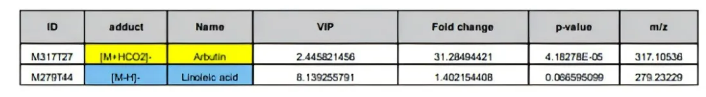
表1 差异代谢物示例表格
注:ID表示代谢物在质谱检测中的编号;adduct表示化合物的加合离子信息;Name表示代谢物的名称;VIP表示变量投影重要度,值越大,表示越重要;FC表示差异倍数;p value表示显著性分析的p值,p值越小,表示差异越显著;m/z表示质荷比。
到目前为止,我们已经确定了代谢组学研究中的关键差异代谢物,接下来的差异分析将在下一期报告中详细呈现。本期的非靶向代谢组报告解读到此结束,期待下次再见~