
01.什么是基因融合
基因融合是指两个基因的部分或全部序列相互融合为一个新的基因的过程。一定比例的融合基因会被转录成嵌合融合转录物,这些嵌合融合转录物可以通过保留其亲本基因的阅读框架形成融合蛋白,可以影响亲本基因表达或作为长的非编码嵌合RNA发挥作用。
一般来说,基因融合是指基因组层面的融合。但转录组层面也可能发生融合,主要是由于两个不同基因转录产生的RNA,由于某种原因融合在了一起,形成新的融合RNA,该RNA可能编码蛋白,也可能为非编码。而基因组层面产生的融合基因,根据融合的情况,可能表达,也可能不表达(如破坏了启动子区域或其他原因)。

▲融合基因示意图
02.融合基因的产生机制
由于染色体层的重排或转录过程中的错误剪接所造成的。在DNA层面上的重排主要包括了6种形式:
染色体内的易位和染色体间的易位;
大片段的插入;
大片段缺失;
串联重复;
倒置;
染色体碎裂导致的复杂重排。
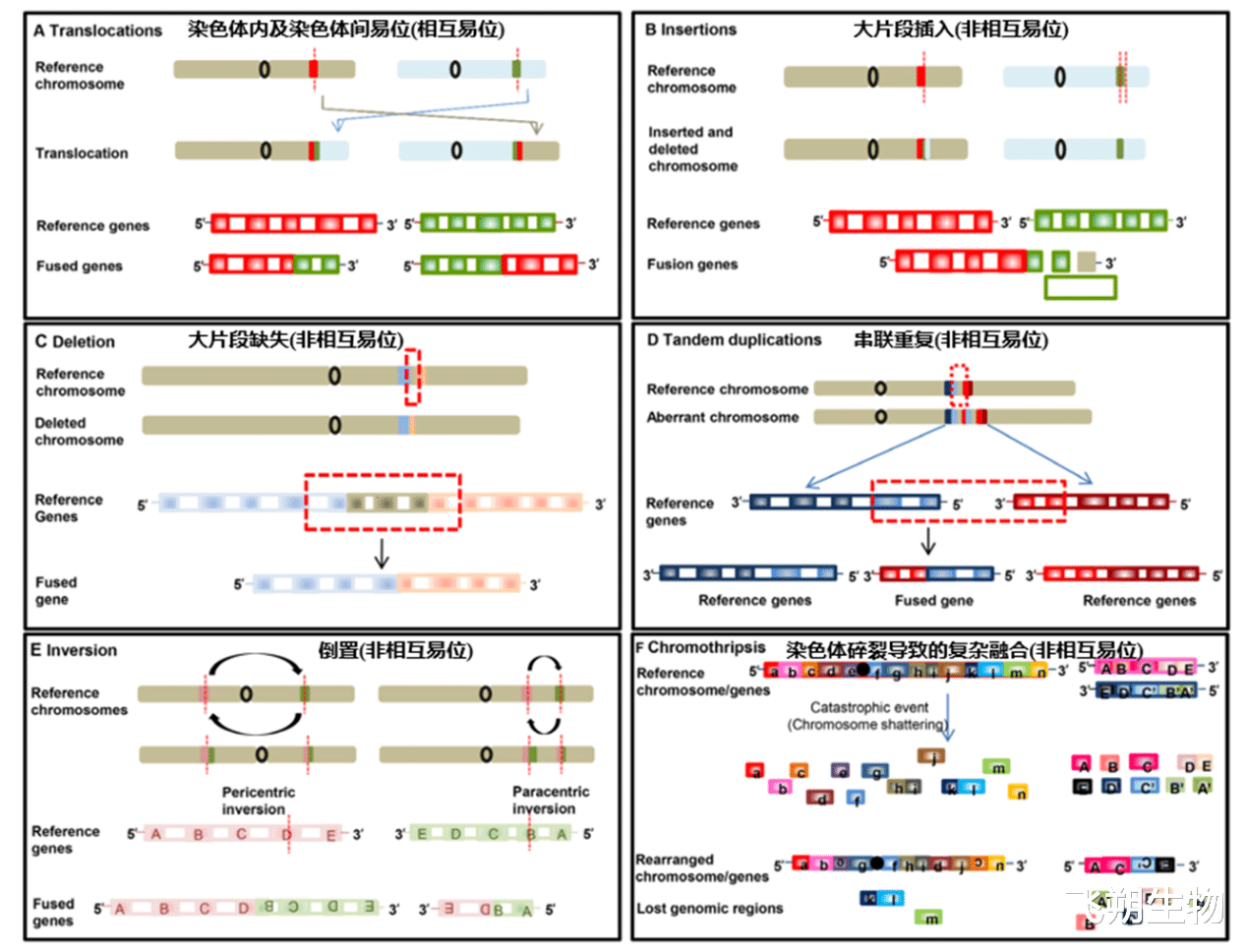
▲DNA层面上的重排形式
在RNA层面上,转录过程中的“通读事件”也可能产生融合蛋白。当RNA聚合酶不能正确终止一个基因末端的转录,并且由于异常剪接而继续转录到下一个基因末端时,产生了嵌合转录子称之为“通读事件”,并不涉及基因组物质的重排(DNA重排)即可产生融合转录子。
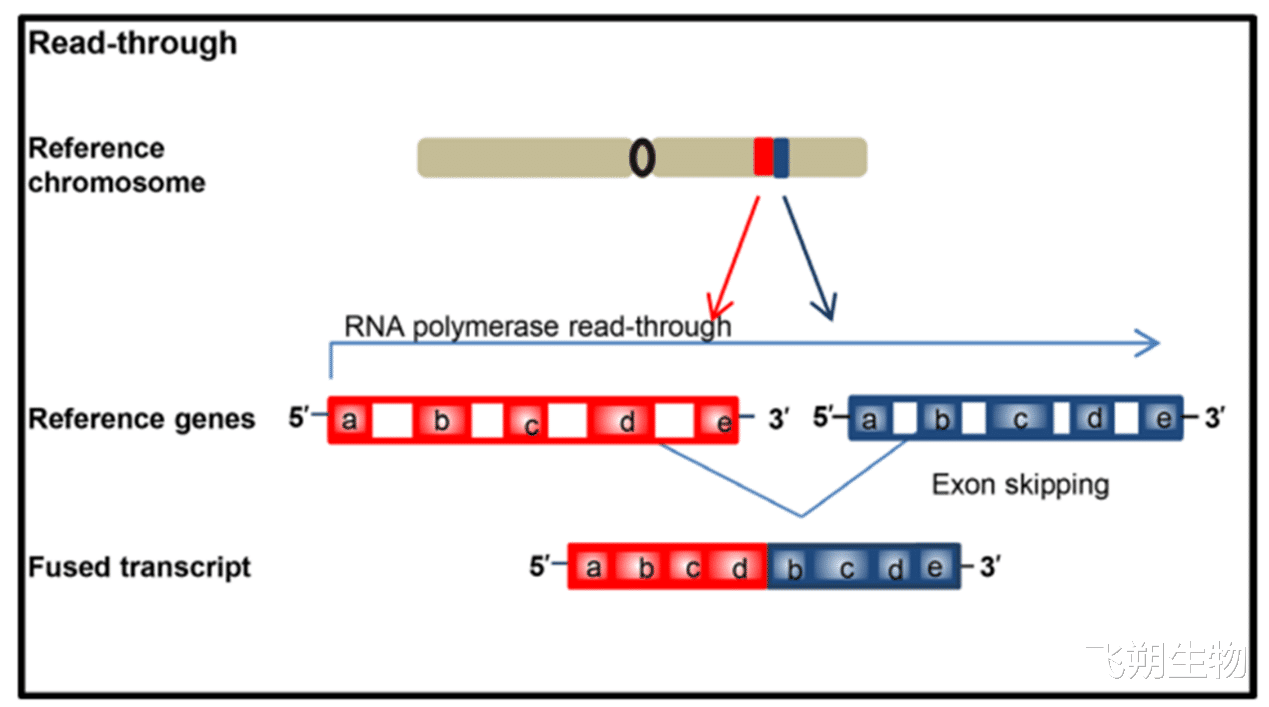
▲转录过程中的“通读事件”
03.融合基因常见检测方法
FISH
通过荧光标记的探针与细胞核内的DNA 靶序列杂交,并在荧光显微镜下观察分析基因扩增或融合变异的一种分子检测技术,一般被认为是检测基因扩增和融合的“金标准”,但也存在一定的局限性。如并非所有检测到的融合均会产生可表达的融合RNA,分离探针可能会遗漏较小的染色体内重排导致假阴性结果,无法确定和区分不同的融合基因变异亚型等。
IHC
利用抗原抗体反应和化学显色原理,检测组织或细胞学切片中特定蛋白表达的技术。IHC检测灵敏度高、成本低、检测时间短、易于自动化,但其准确性取决于检测的融合蛋白是否有经过验证的抗体,如Ventana‑D5F3 IHC检测肺腺癌患者ALK 融合的准确性高,而 ROS1和NTRK阳性IHC结果尚需其他检测方法验证。
RT-PCR
在RNA水平检测融合RNA,灵敏度高、检测时间短,但仅限于检测引物设计范围内的已知融合,无法检出未知融合。此外,该方法检测结果的准确性高度依赖标本RNA的质量。
靶向二代测序
通过大规模平行测序的方法,对引物或探针设计范围内的区域同时进行多个融合基因检测。根据核酸投入物的不同,靶向二代测序可在DNA或RNA水平检测基因融合。
根据中心法则,以DNA为模板通过转录和剪接加工产生成熟的RNA。在DNA水平上,融合断点位置通常发生在较长的内含子区域,且融合的断裂点不同患者可能不同,故用传统的PCR直接扩增断裂点是不易实现的,采用NGS的方法设计探针去抓取断裂点是一种可行的检测方法,但从DNA水平检测融合基因不可避免存在如下局限性和挑战:
1.融合基因的断点一般在内含子区域,而融合基因的内含子又特别冗长,因此,如果基于DNA-based的检测方法就要在内含子区域铺设大量的探针,探针设计难度大。
2.大量探针也会导致下机数据量过大(增加实验成本,加大生信分析难度)。
3.大量探针仍会存在覆盖盲点,导致融合漏检。
4.DNA层面出现融合不一定能转录成mRNA进而翻译成蛋白质,进而不能激活下游MAPK/mTOR通路,而RNA则相对更接近翻译成为蛋白质。
相比DNA水平,RNA水平上融合基因表现为前后两个基因外显子之间的衔接,融合点相对固定。这一特征为精准设计探针或引物提供了先天优势。因此,根据融合基因序列特点,在RNA水平上检测融合基因比DNA水平更易实现。
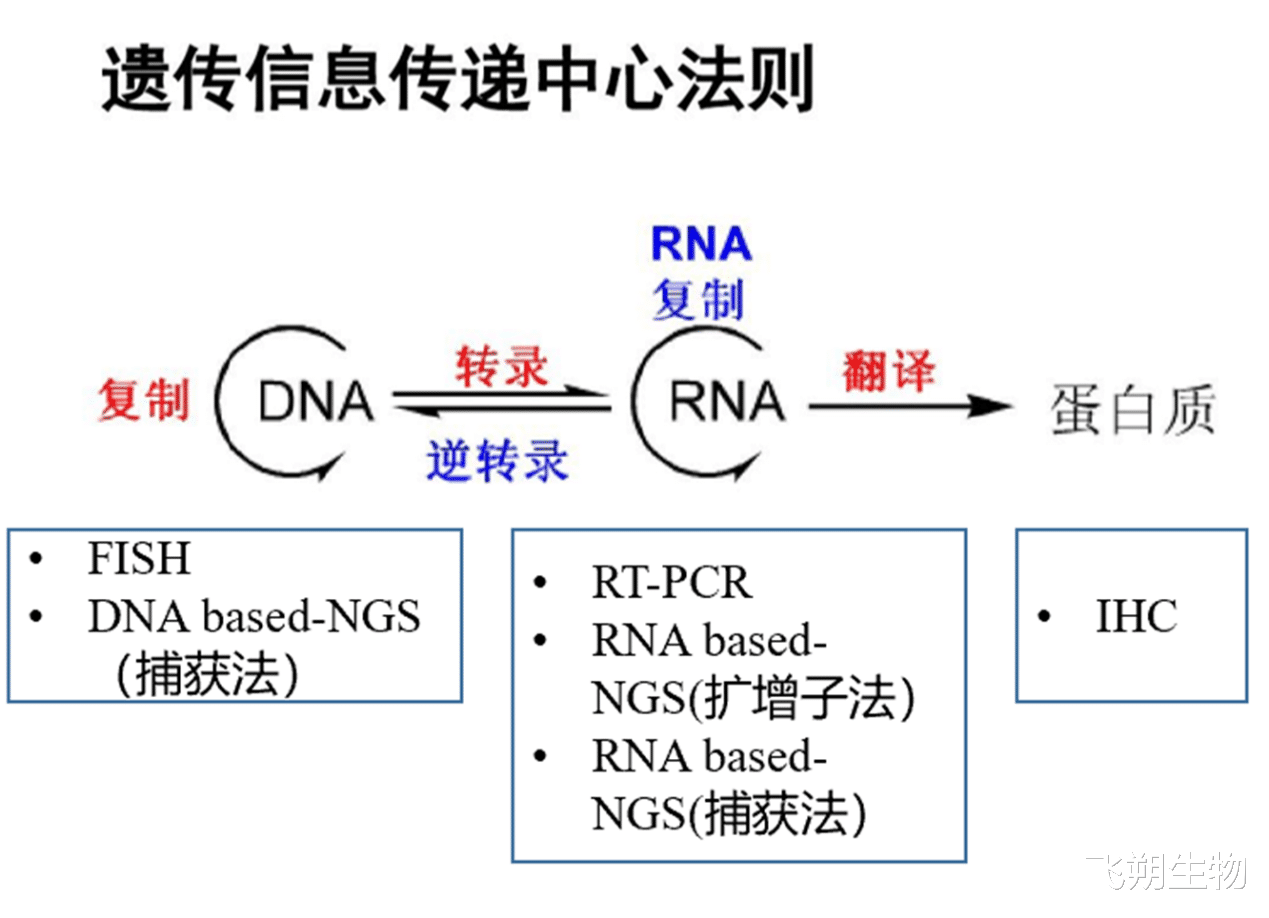
▲融合基因常见的检测方法
04.融合基因检测:DNA vs RNA
DNA-based NGS
原理:对可能发生融合的区域设计探针进行覆盖,杂交捕获得到目的片段进行测序分析。
通过杂交捕获建库,仅使用DNA就可同时对多个肿瘤相关基因的突变、扩增、融合及肿瘤突变负荷(TMB)、微卫星不稳定状态进行检测,极大地节约了标本量,并且能够在DNA水平确定基因融合的断点位置和融合伴侣,检出已知和未知的基因融合。DNA-based NGS测序是基于肿瘤细胞来源的DNA分子进行检测。DNA分子由螺旋的双链组成,相对于RNA更加稳定。因此,DNA-based NGS的组织学/细胞学样本,包括新鲜组织、FFPE、胸水等,均可满足检测质控要求。在组织样本难以获取或样本量不足时,还可以通过液体活检提取ctDNA进行测序,而这是RNA-based NGS无法实现的。但是,DNA二代测序检测基因融合受肿瘤细胞含量、标本DNA质量、捕获探针覆盖度及DNA层面复杂基因变异等影响,可能会出现漏检。另外,随着DNA二代测序在临床分子检测中的广泛应用,越来越多携带罕见伴侣的激酶融合被发现,但某些罕见融合并不能产生有功能的融合RNA/蛋白。
RNA-based NGS
RNA二代测序可以通过多重PCR扩增、锚定多重PCR或杂交捕获建库在RNA水平检测融合转录本。与DNA二代测序需要对外显子和内含子区均进行探针捕获相比,RNA测序仅需要针对外显子区设计引物或探针,相对简单、经济,不受DNA层面复杂融合的影响,且RNA二代测序能直接真实地反映转录水平融合的表达情况及融合伴侣基因类型。
但是,RNA-based NGS检测样本要求相对较高。从样本质量上讲,由于存在核糖核酸酶且RNA是单链结构,非常容易降解,因此,新鲜组织是做RNA-based NGS检测的首选。有研究表明,高达50%的存档FFPE样本,特别是较旧的样本,不能满足RNA建库和测序的要求,可能无法通过测序前的质量控制。目前尚不推荐从液体活检样本中提取RNA进行肿瘤融合基因检测,这也限制了RNA-based NGS检测的应用。
纽约纪念斯隆凯特琳癌症研究中心(MSKCC)的一项真实世界肺腺癌大队列研究和一项中国人群的研究中均通过RNA-based NGS在DNA NGS检测阴性的非小细胞肺癌样本中检出了10%左右的靶点基因融合或MET跳突,相比DNA水平,RNA水平上融合基因表现为前后两个基因外显子之间的衔接,不受内含子的影响,融合点相对固定,这一特征为精准设计探针或引物提供了先天优势,也是RNA-based NGS比DNA-based NGS检测融合基因更具优势的原因之一。

▲纽约纪念斯隆凯特琳癌症研究中心研究

▲中国人群研究

▲NGS检测基因融合对比
05.总结
相比DNA-based NGS,RNA-based NGS不受内含子影响,可提升融合基因的检出率。融合基因DNA和RNA水平上的结构特征决定了融合基因检测,RNA水平远比DNA水平更敏感,更准确,但是样本要求较高。
DNA-based NGS对样本要求相对较低,可联合其他biomarker共同检测,同时DNA层面可检出的罕见融合伴侣、外显子断点融合及基因间融合,但需在RNA或蛋白水平进一步验证。
在临床应用中,全面准确地肿瘤靶基因检测需要临床实践经验的积累以及不断的总结验证。基于检测方法学的局限性和检测标本的复杂性,多平台联合应用或补充有益于肿瘤患者的精准诊治。
参考文献
[1]Oncotarget. 2019 Mar 12;10(21):2095-2111.
[2]Genome Biol. 2020 Jul 6;21(1):166.
[3]Comput Math Methods Med. 2015:2015:912742
[4]DOI:10.3760/cma.j.cn112151-20221111-00946
[5]Noncoding RNA. 2021 Feb 4;7(1):10
[6]https://doi.org/10.1200/JCO.2024.42.16_suppl.e2004
[7]Clin Cancer Res. 2019 Aug 1;25(15):4712-4722
[8]J Thorac Oncol. 2021 Mar;16(3):404-418.
[9 ]DOI: 10.3779/j.issn.1009-3419.2023.102.43
声明:本文仅用于分享,如涉及版权等问题,请尽快联系我们,我们第一时间更正,谢谢!