导语
阐明作物驯化对于深入了解进化和作物改良过程至关重要。亚洲水稻是世界上最重要的主食之一,同时也是生物学研究的绝佳模型,因此人们对亚洲水稻进行了广泛的研究。然而,亚洲水稻的两个主要驯化品种群(粳稻和籼稻)是独立驯化还是仅驯化一次这一根本问题仍未解决,主要有两种不同的假说(多重驯化假说和单一驯化假说)。争论的关键在于粳稻和籼稻共有的驯化等位基因是来自一个野生品系还是多个野生品系。近日,有学者在全基因组水平上对水稻的驯化历史进行了广泛的分析,证明了亚洲水稻的驯化始于不同地区的不同野生谱系,然后通过不同品种群体之间的持续选择和有益等位基因的交换来完成。相关研究成果以题为“Multiple domestications of Asian rice”发表在《Nature plants》。
文章信息

文献标题:Multiple domestications of Asian rice
发表期刊:Nature plants
影响因子:18.0 (Q1)
发表时间:2023.07.04
研究思路
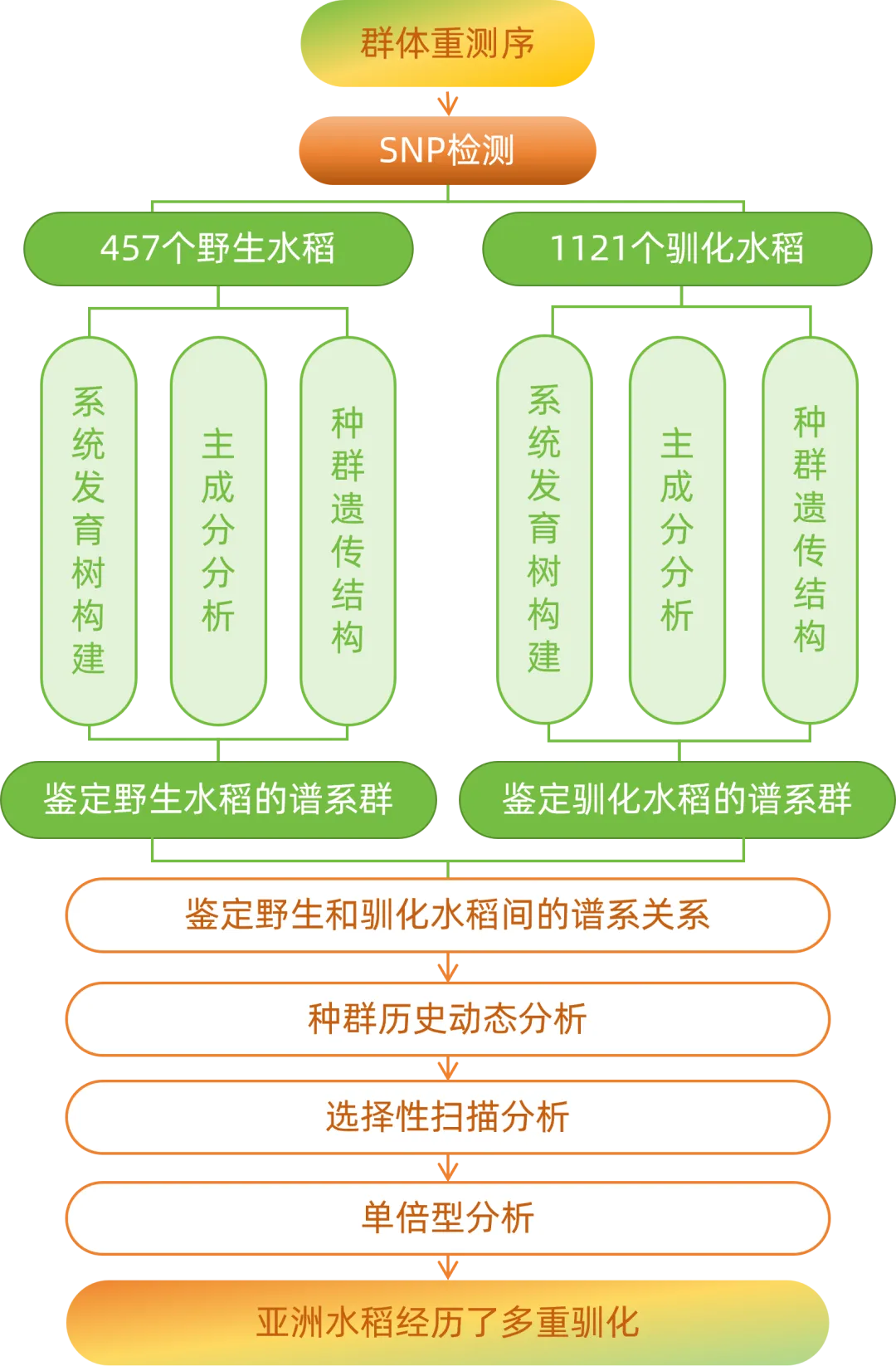
研究结果
1. 野生和栽培材料的鉴定和序列分析
根据15个表型特征,将野生或驯化品种进行了划分,并对那些在最初划分时被错误标注的品种进行了重新分类。在形态学验证的基础上,对422个野生样本(245个O. rufipogon和177个O. nivara)进行了重新测序,平均每个样本测序深度为11.74 X。还对37个驯化水稻样本进行了重测序,其中包括14个代表rayada组的样本。将这些数据与已发表的野生和驯化水稻重测序数据集结合起来,形成了一个包含1,578条全基因组序列的完整数据集,包含457份野生水稻样本和来自25个国家的1,121份驯化水稻样本。以水稻Nipponbare的基因组为参照进行比对,共鉴定出约1,720万个SNPs。
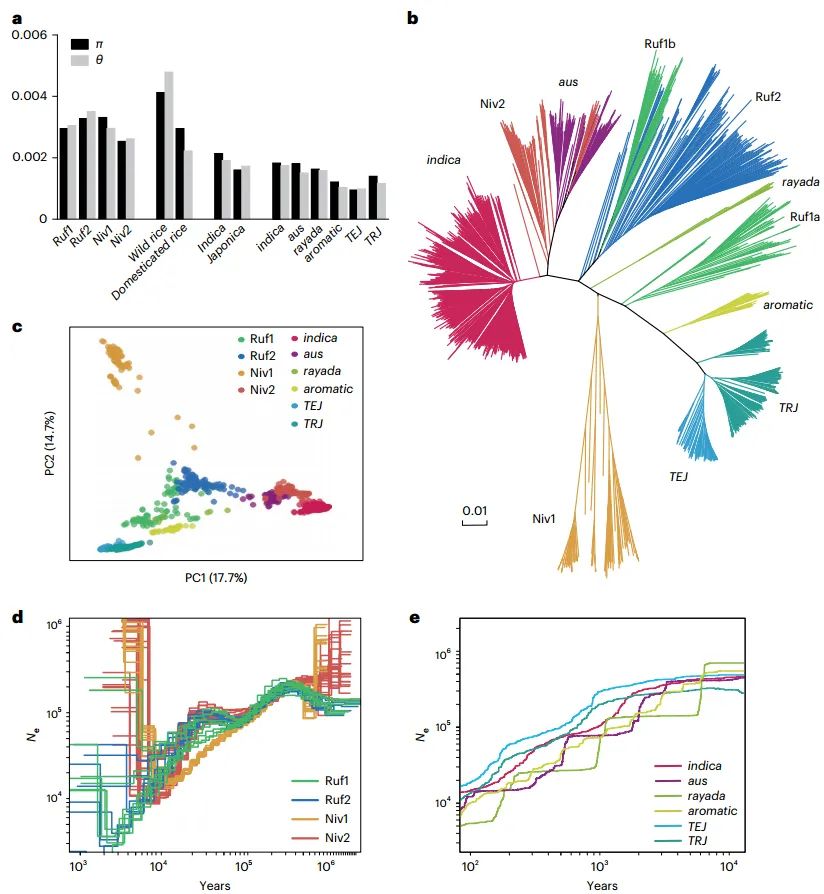
2. 野生水稻和驯化水稻的强遗传结构
为了评估种群遗传结构,使用邻接树(NJ)、主成分分析(PCA)和ADMIXTURE分析了所有457个野生水稻的基因组。这些分析确定了四个主要谱系,包括两个O. rufipogon谱系(Ruf1和Ruf2)和两个O. nivara谱系(Niv1和Niv2)。值得注意的是,两个O. nivara系(Niv1和Niv2)并没有形成一个单系群,而是与不同的O. rufipogon系(Ruf1和Ruf2)关系更为密切。
对驯化水稻的种群遗传学分析显示,NJ树形成了两个独立的单系群。籼稻Indica亚种包括两个驯化品种群(indica和aus),而粳稻Japonica包括四个驯化品种群(aromatic、rayada、温带japonica-TRJ和热带japonica-TEJ)。同样,在K = 2时,ADMIXTURE也检测到两个亚种相对应的两个主要群组,与PCA结果一致。值得注意的是,三个次要驯化品种群(aus、aromatic和rayada)在基因上是不同的,这表明尽管它们在南亚和东南亚有限的地区种植,但仍值得进一步关注。

3. 与驯化相关的种群动态
核苷酸多样性结果显示驯化水稻的多样性(θ=0.0026;π=0.0033)低于野生水稻(θ = 0.0053;π = 0.0046)。引人注目的是,三个次要驯化品种群(aus、rayada和aromatic)的多样性水平接近甚至高于两个粳稻群,表明它们有可能像前人研究中所说的那样是分开驯化的。然后,利用全基因组SNP重建了野生和驯化水稻谱系之间的种群结构。NJ树和PCA都表明,驯化水稻并没有形成一个单系群,而是分成了几个不同的类群,这些类群与不同的野生水稻谱系关系密切,这与之前基于全基因组中性标记的研究结果一致。具体来说,籼稻(indica + aus)与南亚和东南亚的一个O. nivara亚系(Niv2)形成一个群,而三个粳稻群(aromatic、TEJ和TRJ)与中国南方的一个O. rufipogon亚系(Ruf1a)聚类,这表明南亚北部的O. rufipogon亚系(Ruf1b)没有直接参与水稻驯化。
4. 选择性扫描区和受选择基因分析
在100kb滑动窗口以10kb的步长扫描基因组,确定了98个选择性扫描区域(PSRs)。利用每个PSR中的SNPs重建四个野生和两个驯化种群的NJ树,鉴定出71个PSRs,且粳稻和籼稻形成了一个单系群,这表明72.4%的PSR只从O. rufipogon系或O. nivara系起源过一次。将这些PSRs定义为单源选择性扫描区域(SORs)。这71个SORs占参考基因组的6.5%,分布在所有染色体上。不出所料,在SORs中发现了许多具有功能特征的驯化基因,包括sh4、PROG1、Rc等。根据水稻驯化种群的不同野生姐妹群,将71个SOR树分为七类,在77.4%的SOR树中,Ruf1是驯化种群的姐妹群,而在14.1%的SOR树中,Niv1和Niv2是野生姐妹群。这些观察结果表明,O. rufipogon和O. nivara都为水稻种质提供了驯化等位基因。
比较了基于区域和基于基因的扫描结果,得出了两点结论。首先,1882个PSG中有1400个(74.4%)包含在98个PSR中。同样,993个SOGs中有791个(79.7%)在71个SORs中被发现。第二个出乎意料的发现是,在71个SORs中,有6个不同起源(即来自O. rufipogon或O. nivara)的SOGs分布在一起。为了进一步探究这一现象,分析了两个源自不同野生祖先的SOR(SOR33和SOR48)。在LABA1和LCBK2所在的SOR33中,Niv1或Niv2与LABA1树上的驯化品种群是姐妹关系,与SOR33树一致,而Ruf1与LCBK2树上的驯化品种群关系最为密切,与SOR33树不同。在SOR48中,PROG1和GLO4邻近共存,在PROG1树上,Ruf1与栽培品种组为姐妹关系,而在GLO4树上,Niv1与栽培品种组关系最近。这些观察结果表明,一个选定的区域可能由多个片段/基因镶嵌而成,这些片段/基因体现了不同的进化历史。

5. 单倍型分析显示多次驯化
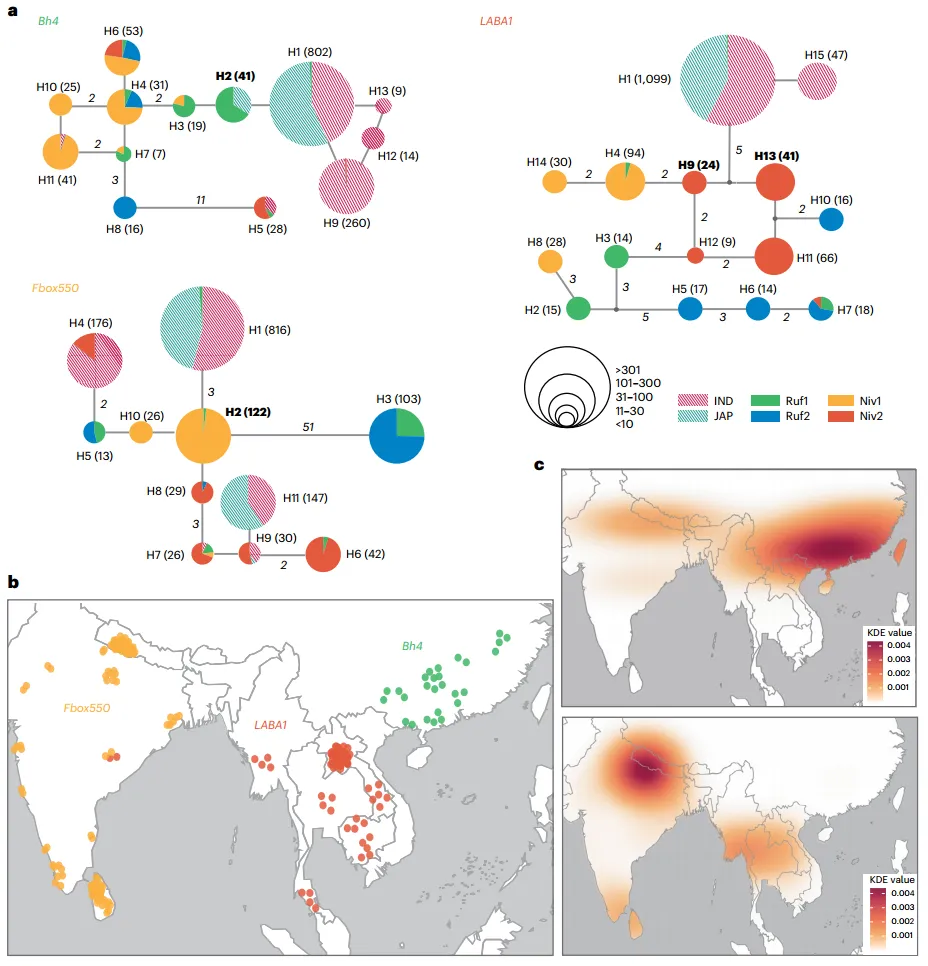
研究总结
该研究收集了1,578个重测序的基因组数据集,其中包括来自野生水稻整个地理分布范围的样本。检测到了993个选择性基因,这些基因构建的系统发生树上,粳稻和籼稻形成了一个单系群,且驯化等位基因只起源于粳稻或籼稻。重要的是,大多数所选基因(约80%)的驯化等位基因源自中国的野生稻,但相当少数所选基因(约20%)的驯化等位基因源自南亚和东南亚的野生稻,这表明亚洲水稻的驯化事件是独立的。
参考文献
Jing CY, Zhang FM, Wang XH, Wang MX, Zhou L, Cai Z, Han JD, Geng MF, Yu WH, Jiao ZH, Huang L, Liu R, Zheng XM, Meng QL, Ren NN, Zhang HX, Du YS, Wang X, Qiang CG, Zou XH, Gaut BS, Ge S. Multiple domestications of Asian rice. Nat Plants. 2023 Aug;9(8):1221-1235. doi: 10.1038/s41477-023-01476-z. Epub 2023 Aug 7. PMID: 37550371.