引言
转录因子(TF)是能够与顺式作用元件特异性相互作用,并对基因转录进行激活或抑制的DNA结合蛋白;转录因子结合位点(TFBS)是转录因子调节基因表达时,与基因模板链发生结合的特定区域(5-20bp);一个TF可以同时调控多个基因,其在不同基因上的TFBS具有一定的保守性。
通过全基因组范围内鉴定转录因子结合位点,可以更全面地了解转录因子是如何在基因组上选择特定的位置进行结合,从而实现对基因表达的精细调控,这对于揭示转录因子在各种生物过程中的作用机制具有重要作用。DAP-seq可以通过结合DNA-体外蛋白相互作用实验和高通量测序,能够高效精准地鉴定TFBS,DAP-seq通过在体外环境中重建DNA-蛋白相互作用,使得实验操作简便并能排除细胞环境对结合事件的干扰(O’Malley et al.2016)。
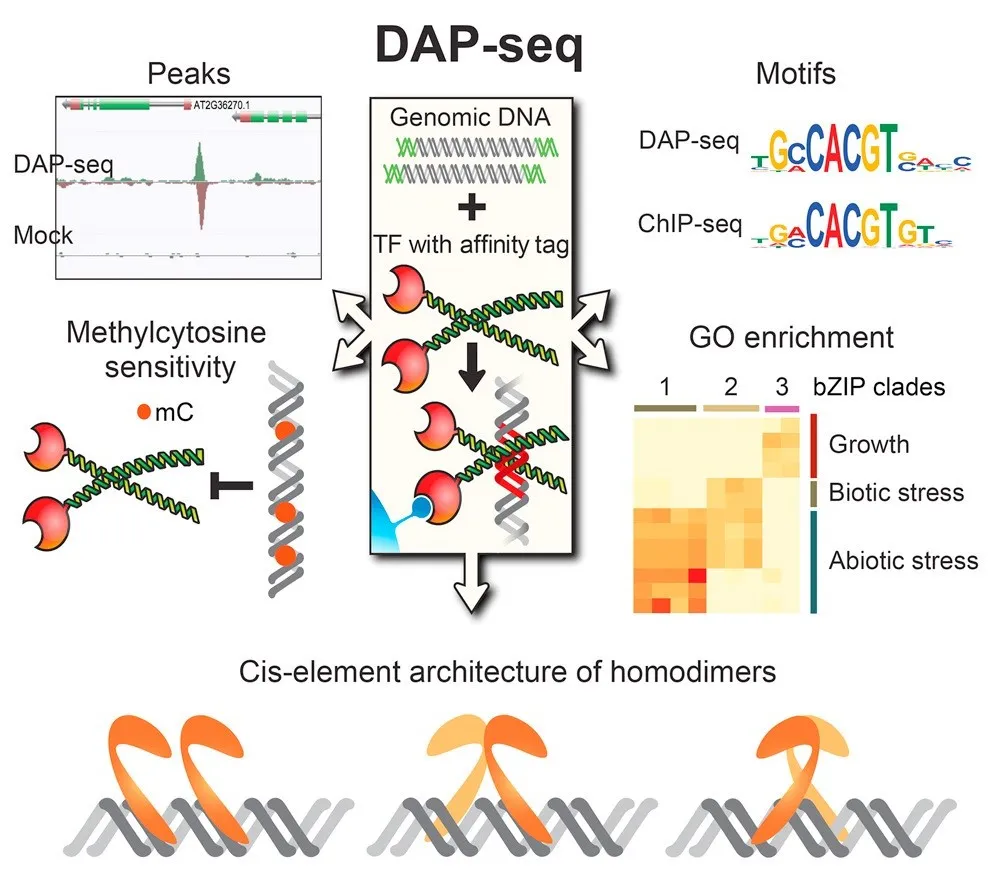
Fig1:DAP-seq技术原理
DAP-seq技术介绍
DAP-seq(DNA亲和纯化测序),是一种高效的体外鉴定转录因子结合位点(TFBS)的方法;该方法是通过将体外表达的转录因子(TF)和基因组DNA进行亲和纯化,然后将TF捕捉到的DNA片段进行高通量测序,通过对高通量测序得到的序列数据进行分析确定感兴趣的TFBS(Bartlett et al.2017)。
技术优缺点:DAP-seq技术采用体外表达标签蛋白(多为TF),将纯化蛋白与目标基因组DNA片段孵育进而确定蛋白结合序列片段,因此不需要特异性抗体,不需要转基因,可用于没有稳定遗传转化体系的物种和没有特异性抗体的目标蛋白;但缺点是DAP-seq是体外实验,而且是结合片段的基因组DNA(已经没有了体内的空间和结构信息),因此无法真实反映体内转录因子与DNA的互作。
DAP-seq技术流程
1) 基因组文库构建:首先,从样品中提取高质量的基因组 DNA,并将其进行物理或酶切片段化。随后,通过连接基于 Illumina 平台的测序接头,构建基因组 DNA 文库。这一步骤确保了 DNA 片段的端修复、加 A 尾及连接测序接头的完整性,为后续的高通量测序奠定基础。
2) 转录因子体外表达纯化:将编码目标转录因子的 CDS(Coding DNA Sequence)序列克隆到带有亲和标签(如 Halo Tag)的表达载体中。构建完成后,将其转入合适的表达系统中进行体外蛋白表达。通过这种方式,我们可以获得融合了亲和标签的转录因子蛋白,以便后续的亲和纯化和分析。
3) 亲和纯化及测序:纯化表达的转录因子-亲和标签融合蛋白后,将其与预先构建的基因组 DNA 文库共同孵育。融合蛋白中的转录因子能够特异性地结合 DNA 片段上的目标序列。利用 Halo Tag 特异性磁珠进行亲和纯化,从混合物中提取出结合了目标 DNA 的蛋白-DNA 复合物。然后,通过 PCR 扩增捕获的 DNA 片段,最终使用 Illumina 高通量测序平台进行测序,以获得高质量的序列数据。
4) 数据分析:将获得的测序读数与参考基因组进行比对分析,识别出目标转录因子的潜在结合位点。通过生物信息学分析,可以进一步了解这些结合位点在基因组中的分布及其在调控基因表达中的潜在功能。这样可以帮助我们深入理解转录因子的生物学作用及其调控机制。
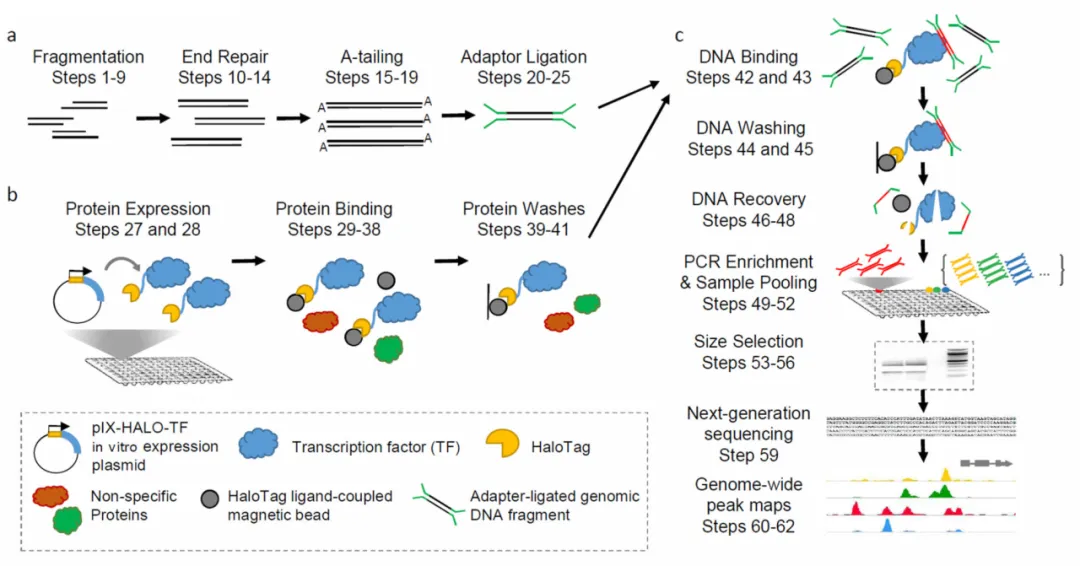
Fig2:DAP-seq技术流程
经典案例
案例一:解码玉米胚乳分化的基因调控网络
研究使用ampDAP-seq技术获得了161个转录因子的基因组结合图谱,与单细胞转录组数据整合构建了一个高置信度的基因调控网络,通过进一步的调控网络分析,研究筛选出了细胞类型特异的关键调控因子,并验证了其在BETL细胞发育中的租用,为农业研究提供了针对性的调控网络数据(Yuan et al.2024)。
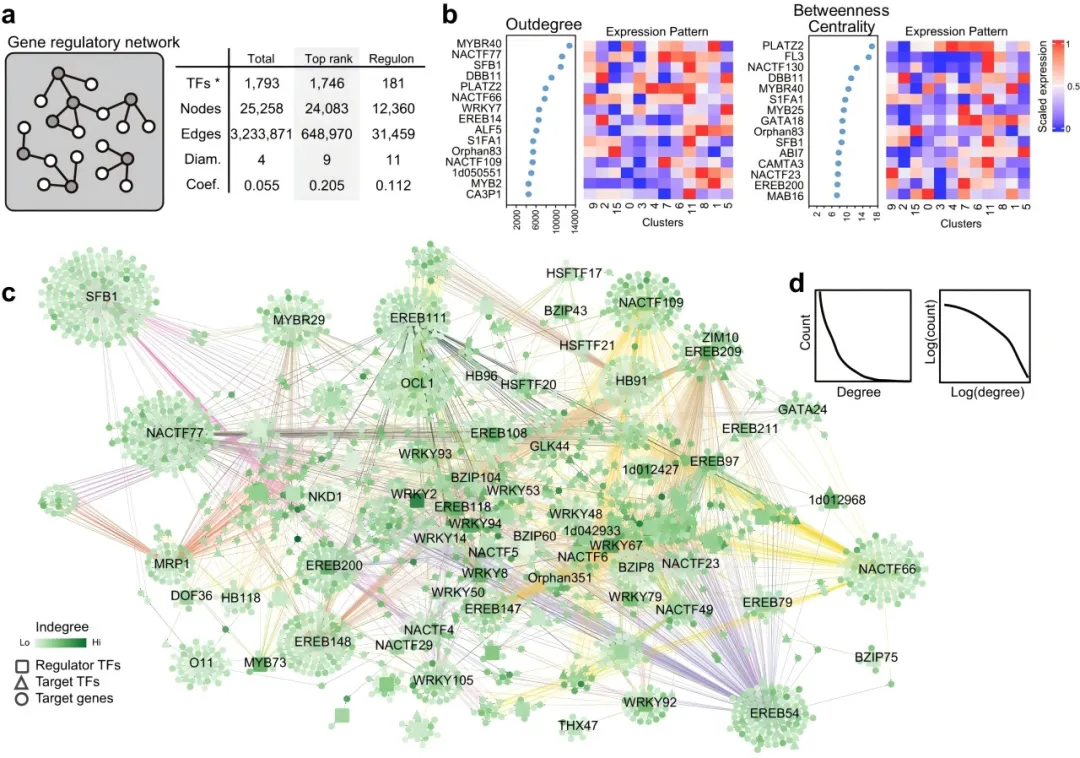
Fig3:高置信度玉米调控网络的重建
案例二:OsMYB8的自然变异赋予了籼稻和粳稻亚种之间的日间开花时间差异
研究将OsMYB8确定为水稻日间开花时间(DFOT)的关键调节因子,通过DAP-seq流程分析并实验验证证实了OsMYB8诱导JA-Ile合成酶OsJAR1的转录,从而调节细胞渗透压和细胞壁重塑相关基因的表达,促进小花开放;研究结果揭示了OsMYB8-OsJAR1模块,该模块调控籼稻和粳稻的差异DFOT,为早期DFOT粳稻杂交种提供了策略,以促进籼稻-粳稻杂交种的育种(Gou et al. 2024)
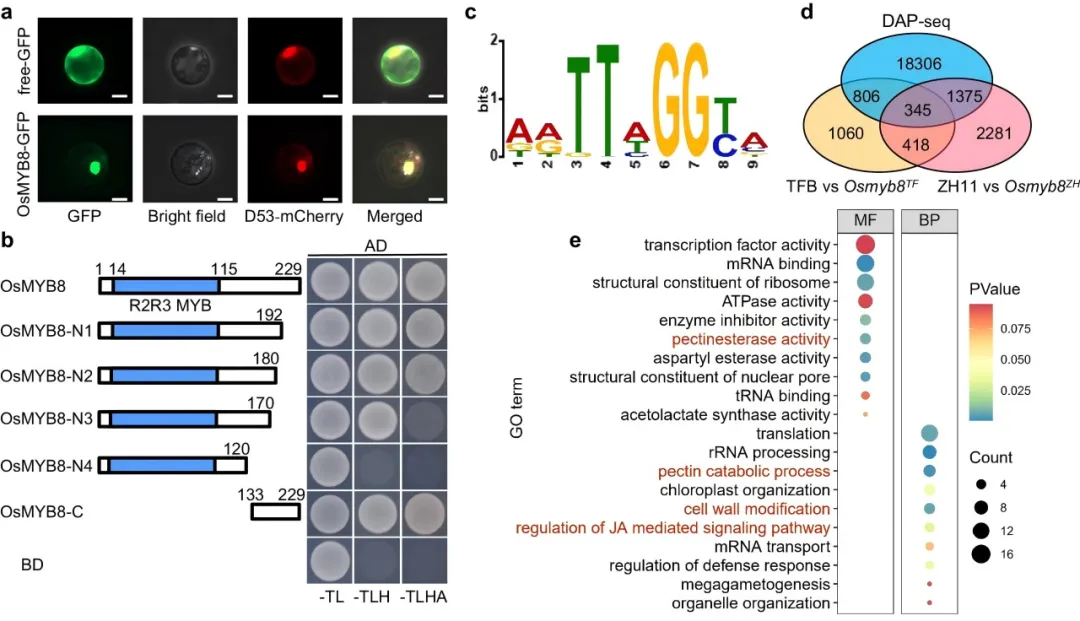
Fig4:OsMYB8转录因子全基因组靶标的鉴定
参考文献
Bartlett, Anna, et al. ‘Mapping Genome-Wide Transcription-Factor Binding Sites Using DAP-Seq’. Nature Protocols, vol. 12, no. 8, Aug. 2017, pp. 1659–72. 14.8, https://doi.org/10.1038/nprot.2017.055.O’Malley, Ronan C., et al. ‘Cistrome and Epicistrome Features Shape the Regulatory DNA Landscape’. Cell, vol. 165, no. 5, May 2016, pp. 1280–92. 64.5, https://doi.org/10.1016/j.cell.2016.04.038.Yuan, Yue, et al. ‘Decoding the Gene Regulatory Network of Endosperm Differentiation in Maize’. Nature Communications, vol. 15, no. 1, Jan. 2024, p. 34. 16.6, https://doi.org/10.1038/s41467-023-44369-7.Gou, Yajun, et al. ‘Natural Variation in OsMYB8 Confers Diurnal Floret Opening Time Divergence between Indica and Japonica Subspecies’. Nature Communications, vol. 15, no. 1, Mar. 2024, p. 2262. 16.6, https://doi.org/10.1038/s41467-024-46579-z.