骨肉瘤(Osteosarcoma,OS)是最常见的原发性恶性骨肿瘤,主要在青少对时期发病,在所有儿童癌症中占比约2.4%。该病最常见于10至30岁的人群中,男性患者略多于女性。流行病学基因解码发现,非裔美国人、亚裔/太平洋岛民和西班牙裔人群患骨肉瘤的比例高于白人。
佳学基因致力于揭示骨肉瘤的具体发病机制,已有多项基因解码指出若干流行病学因素可能与其发病风险上升密切相关。例如,遗传性视网膜母细胞瘤、Li-Fraumeni 综合征、Rothmund-Thompson 综合征以及Bloom和Werner综合征患者被认为患骨肉瘤的风险显著增高。此外,骨Paget病、骨纤维发育不良和放射线暴露等也被认为是与老对人骨肉瘤发病相关的诱发因素。有趣的是,相比一般人群,身材较高的人被观察到患骨肉瘤的概率更大。
骨肉瘤包含多个亚型,其中大部分属于高级别,并表现出高度侵袭性,高度转移性。尽管治疗手段不断进步,但整体预后仍不甚理想。由于个体间基因差异的存在,化疗药物的疗效和不良反应呈显著异质性。佳学基因之所以深入探究这些差异背后的基因信息基础,是希望推动骨肉瘤治疗的进一步发展。
单核苷酸多态性(SNP)是指基因组中某个位点上的单个碱基变异,存在于外显子、内含子或调控区域。SNP常与基因表达水平变化、癌症风险和预后相关,并且常常处于与致病位点的连锁不平衡状态。目前已有基因解码指出多个与骨肉瘤预后密切相关的基因和代谢通路,包括ABCB1、GSTP2、VEGFD、GRM4及一些参与DNA修复的关键酶。佳学基因已总结了这些SNP在预测骨肉瘤治疗结果方面的基因解码证据。
基因解码还发现了一些在细胞生长与抑癌功能方面关键基因中的常见SNP,提出其可能影响骨肉瘤易感性,例如CTLA-4、ERCC2与TP53等。但这些基因解码通常受限于样本量较小,结果之间缺乏一致性,难以形成确切结论。此外,在胃癌等疾病中,已有基因解码显示不同族群之间的SNP与疾病风险关联存在差异,这也提示佳学基因在合并不同种族的数据分析时应格外谨慎。
虽然已有若干系统评价探索了某些特定基因或单个SNP与骨肉瘤之间的关系,但迄今为止,关于所有与骨肉瘤相关SNP的系统整合分析仍较为稀缺。这一现状突显了对该领域进行更全面基因解码的必要性。
佳学基因的系统评价工作正是为填补这一空白而展开,旨在全面梳理和评估与骨肉瘤风险相关的SNP证据。虽然过去的荟萃分析已对部分候选基因进行了探讨,但涵盖所有潜在相关SNP的系统性综述,特别是结合种族因素进行分析的基因解码仍非常有限。因此,该评价不仅着眼于骨肉瘤相关SNP的全面识别与分析,还高度关注种族差异对骨肉瘤遗传易感性的影响。这一工作将为深入理解骨肉瘤的遗传机制提供重要基础,也有望为临床个体化诊疗方案的制定提供理论支持。
骨肉瘤的易感基因解码是如何进行的?主分析和留一分析的结果本荟萃分析仅纳入 48 项遗传关联基因解码。针对 23 个不同基因的 37 种遗传变异(35 个 SNP 和 2 个 DELINS),至少有两项独立基因解码可用,这使佳学基因检测能够进行 183 项荟萃分析。佳学基因检测使用随机效应模型计算了荟萃分析中包括的 35 种遗传变异的汇总优势比 (OR) 和 95% 置信度。在主要分析中,8 个基因(CTLA-4、ERCC3、IL-8、PRCKG、RECQL5、TNF-α、XRCC3 和 VEGF)的 12 种变异的汇总 OR 与骨肉瘤风险显著相关。而在敏感性分析中,MDM2的2个SNP 与骨肉瘤风险显著相关。佳学基因检测计算了 5 种遗传模型(纯合、杂合、显性、隐性和等位基因)的汇总 OR。佳学基因检测还分别报告了亚洲人和高加索人种的估计值,以基因解码种族的影响。表1显示了合并分析或其中一个人群中与骨肉瘤相关的基因变异。
表 1.与骨肉瘤相关的基因变异。SNV染色体模型或 (95% 置信区间) P 值I2 %Q值异质性P值病例组与对照组#亚洲基因解码亚裔 OR (95% CI)#基因解码白种人白种人 OR (95% CI)VEGF rs6999476CA 与 CC1.33[1.10;1.61]0.01800.860.83709 对 874 (4)41.33[1.10;1.610AA 与 CC1.89[1.31; 2.72] 0.01201.690.64709 对 874 (4)41.89[1.31; 2.72]0CA + AA 与 CC1.46[1.16; 1.84]0.01401.480.69709 对 874 (4)41.46[1.16; 1.840AA 与 CC + CA1.64[1.24;2.16]0.01101.100.77709 对 874 (4)41.64[1.24;2.16]0C 对比 A1.40[1.16;1.70]0.01101.90.59709 对 874 (4)41.40[1.16;1.70]0VEGF rs30250396CT 与 CC1.15[1.08;1.22]0.000801.240.991913对与2302对(9)91.15[1.08;1.22]0TT 与 CC1.73[1.38;2.16]0.000504.830.771913对与2302对(9)91.73[1.38;2.16]0CT + TT 对抗 CC1.25[1.17;1.33<0.000100.560.991913对与2302对(9)91.25[1.17;1.330TT 与 CC + CT1.61[0.92; 2.82] 0.0975.332.37< 0.00011913对与2302对(9)91.61[0.92; 2.82]0T 与 C1.27[1.18;1.36]<0.000102.830.941913对与2302对(9)91.27[1.18;1.36]0VEGF rs15703606GA 与 AA1.16[0.95;1.42]0.0800.250.88527 比 692 (3)31.16 [0.95; 1.42]0GG 对阵 AA1.44[1.33;1.55]0.002300.020.99527 比 692 (3)31.44[1.33;1.55]0GA + GG 与 AA1.24[0.996;1.54]0.05100.360.83527 比 692 (3)31.24 [0.996; 1.54]0GG 与 AA + GA1.36[1.29;1.43]0.00200.010.99527 比 692 (3)31.36[1.29;1.43]0G 与 A1.24[1.02;1.5]0.04200.470.79527 比 692 (3)31.24[1.02;1.5]0VEGF rs20109636CG 与 CC1.30[1.13;1.50]0.00402.610.861489对与1867对(7)71.30[1.13; 1.50]0GG 对 CC1.56[1.12;2.18]0.0183769.550.151489对与1867对(7)71.56[1.12; 2.18]0CG + GG 对抗 CC1.35[1.094;1.65]0.013136.930.331489对与1867对(7)71.35[1.094;1.65]0GG 对阵 CG + CC1.47 [0.90; 2.50] 0.128539.44< 0.00011489对与1867对(7)71.47 [0.90; 2.50]0G 与 C1.26[1.06;1.49]0.0174410.690.0981489对与1867对(7)71.26[1.06;1.49]0VEGF rs104346GA 与 GG1.08[0.98;1.18]0.1100.920.971167对与1524对(6)61.08[0.98;1.18]0AA 对阵 GG1.21[1.07;1.37]]0.01200.810.981167对与1524对(6)61.21[1.07;1.37]]0GA + AA 对比 GG1.12[1.01;1.21]0.03400.910.961167对与1524对(6)61.12[1.01; 1.21]0AA 对比 GG + GA1.17[1.08;1.27]0.00400410.991167对与1524对(6)61.17[1.08;1.27]0G 与 A1.10[1.04;1.17]0.0100.830.971167对与1524对(6)61.10[1.04; 1.17]0CTLA-4 rs57429092CT 与 CC1.31[0.06;27.42]0.47502.010.16389 比 413(2)21.31[0.06;27.42]0TT 与 CC2.5[1.4;4.4]0.03200.010.91389 比 413(2)22.5[1.4;4.4]0.0320CT + TT 对抗 CC1.41[0.085;23.48]0.3601.90.168389 比 413(2)21.41 [0.085; 23.48]0TT 与 CC + CT2.30[1.71;3.08]0.017470.0010.95389 比 413(2)22.30[1.71;3.08]0T 与 C1.43 [0.178; 11.47] 0.28311.450.23389 比 413(2)21.43 [0.178; 11.47]0CTLA-4 rs2317752GA 与 GG1.02[0.37;2.8]0.94505.960.11693 对 754 (4)31.25 [0.89; 1.75]10.28[0.08;1.001]AA 对阵 GG1.93[0.96;3.91]0.0602.790.43693 对 754 (4)32.24[2.14; 2.34]10.72[0.20;2.59]GA + AA 对比 GG0.69[0.06;7.80]0.669236.54< 0.0001693 对 754 (4)31.40[1.04;1.88]12.40(0.70-8.18)AA 对比 GG + GA0.75[0.5;1.11] 0.102.170.54693 对 754 (4)31.98[1.73;2.26]12.24[1.21;4.15]G 与 A1.1[0.5; 2.4] 0.728722.26< 0.0001693 对 754 (4)31.38[1.20;1.59]11.36[0.87; 2.14]ERCC3 rs415050619CT 与 CC1.26[1.04;1.53]0.0400.020.90522与1047(2)21.26[1.04;1.53]0TT 与 CC1.78[1.20;2.63]0.00400.400.53522与1047(2)21.78[1.20;2.63]0CT + TT 对抗 CC1.35[1.09;1.67]0.00600.10.75522与1047(2)21.35[1.09;1.67]0TT 与 CC + CT1.62[1.11;2.37]0.0100.470.49522与1047(2)21.62[1.11; 2.3]0C 与 T1.33[1.12;1.57]0.00100.330.56522与1047(2)21.33[1.12;1.57]0IL-8 rs40734TA 与 TT1.60[0.99; 2.6] 0.050100.040.85299 比 299 (2)21.60[0.99; 2.6]0AA 对抗 TT1.95[1.53;2.50]0.0200.010.94299 比 299 (2)21.95[1.53;2.50]0TA + AA 对抗 TT1.70[1.05;2.73]0.0500.0010.96299 比 299 (2)21.70[1.05;2.73]0AA 与 TA + TT1.67[1.39;2.01] 0.0200.0010.95299 比 299 (2)21.67[1.39;2.01]0A 与 T1.6[1.03;2.49]0.0500.070.8299 比 299 (2)21.6[1.03;2.49]0IL-6 rs18007957GC 与 GG1.09[0.79;1.51]0.53888.570.003286 对 366 (2)11.57 [1.04; 2.36]10.55[0.31;1.002]CC 对阵 GG1.0[0.72;1.54]0.77856.770.0093286 对 366 (2)11.77 [1.02; 3.07]10.63[0.37;1.10]GC + CC 对抗 GG1.14[0.82;1.59]0.439733.65< 0.0001286 对 366 (2)12.03 [1.37; 3.0]10.51[0.29;1.005]CC 对比 GG + GC1.06[0.002;721.8] 0.9266.770.0093286 对 366 (2)10.45[030;1.001]10.70[0.41;1.21]C 与 G1.09[0.87;1.38]0.50970.95< 0.0001286 对 366 (2)11.76[1.31;2.36]10.45 [0.30;1.004]IL-10 rs18008961AG 与 AA1.23[0.85;1.76]0.27612.60.2340 对 420 (2)11.07 [0.72; 1.59]12.67 [0.95; 7.50]GG 对阵 AA1.92[0.64;5.75] 0.0800.130.72340 对 420 (2)11.85[1.10;3.09]12.3 [0.79; 6.67]AG + GG 对抗 AA1.38[0.99;1.90] 0.05200.880.35340 对 420 (2)11.29 [0.91; 1.82]12.02[0.84;4.89]GG 对阵 AA + AG1.36[0.03; 57.5] 0.48582.390.12340 对 420 (2)11.80[1.10; 3.0]11 [0.57; 1.75]G 与 A1.31[0.48;3.60]0.18400.480.49340 对 420 (2)11.39[1.06;1.81]11.17 [0.79; 1.74]GSTP1 rs169511AG 与 AA1.19[0.79;1.81]0.2101.090.58400 对 783 (3)11.43 [0.93; 2.18]21.08[0.82;1.40]GG 对阵 AA2.09[0.64;6.88]0.1721.12.540.28400 对 783 (3)13.33[1.53;7.30]21.50 [0.60; 3.70]AG + GG 对抗 AA1.30 [0.73 2.31] 0.1810.32.230.33400 对 783 (3)11.66[1.11;2.49]21.1276 [0.99; 1.27]GG 对阵 AA + AG1.91 [0.69; 5.31] 0.1101.980.37400 对 783 (3)12.85 [1.34; 6.05]21.44 [0.52; 3.98]G 与 A1.31[0.78;2.20]0.1503.060.22400 对 783 (3)11.6170[1.19;2.20]21.141[1.05;1.24]PRKCG rs45400619CT 与 TT0.96[0.09;10.29]0.86703.340.07998 对 998 (2)20.96[0.09;10.29]0CC 与 TT1.94[1.69;2.23]0.0100.010.94998 对 998 (2)21.94[1.69;2.23]0CT + CC 对阵 TT1.20[0.34;4.20]0.32151.180.28998 对 998 (2)21.20 [0.34; 4.20]0CC 对阵 CT + TT1.99 [0.82; 4.83] 0.0700.280.60998 对 998 (2)21.99 [0.82; 4.83]0C 与 T1.35[1.11;1.63]0.0300.050.83998 对 998 (2)21.35[1.11; 1.63]0RECQL5 rs82019617TC 与 TT1.31[1.11;1.55]0.0300.010.94397 比 441(2)21.31[1.11;1.55]0CC 与 TT2.52 [1.60; 4.0] < 0.000100.150.70397 比 441(2)22.52 [1.60; 4.0]0TC + CC 对抗 TT1.49[1.03; 2.14] 0.0500.040.84397 比 441(2)21.49[1.03; 2.14]0CC 与 TT + TC2.15[1.41;3.29]0.000400.150.79397 比 441(2)22.15[1.41;3.29]0C 与 T1.44[1.19;1.76]0.000300.110.74397 比 441(2)21.44[1.19;1.76]0TNF-α rs18006296GA 与 GG1.18[0.46;3.05]0.52162.380.30227 比 370(3)11.06[0.56;2.01]21.26 [0.77; 2.06]AA 对阵 GG3.46 [0.40; 29.90] 0.0900.170.67160 对 259 (2)14.0 [1.41; 11.38]12.84[0.83;9.68]GA + AA 对比 GG1.37 [0.50; 3.78] 0.31332.980.23227 比 370(3)11.39 [0.76; 2.53]21.37 [0.86; 2.18]AA 对比 GG + GA3.26 [0.22; 48.28] 0.1100.290.60160 对 259 (2)11.39 [0.76; 2.53]11.37 [0.86; 2.18]A 对 G1.46 [0.54; 3.93] 0.24443.570.17227 比 370(3)11.61[1.04;2.50]21.46 [0.54; 3.93]TNF-α rs 3615256GA 与 GG0.53[0.28;0.98]0.04703.340.07143 对 210 (2)10.80[0.39;1.63]10.17[0.04; 0.76]AA 对阵 GG*不适用GA + AA 与 GG*不适用AA 与 GG + GA*不适用A 对 G0.65[0.38;1.124]0.1244.030.045143 对 210 (2)10.95[0.51;1.76]10.18[0.04;0.8]TP53 rs104252217CG 与 CC0.79[0.34;1.82]0.437512.00.01615 对 844 (4)11.50[1.03;2.20]30.59[0.36;1.001]GG 对 CC1.50[0.66;3.38]0.2131.64.380.22615 对 844 (4)11.89[1.16;3.07]31.16 [0.26; 5.08]CG + GG 对抗 CC0.98[0.49; 1.93] 0.9166.28.900.03615 对 844 (4)11.57[1.10;2.25]30.77[0.40;1.50]GG 对阵 CC + CG1.70[0.70;4.13]0.1502.840.41615 对 844 (4)11.47 [0.96; 2.26]31.91 [0.28; 13.23]G 与 C1.182 [0.74; 1.88] 0.3451.46.180.10615 对 844 (4)11.38[1.09;1.75]31.09[0.47;2.56]XRCC3 rs86153914CT 与 CC1.37[1.34;1.39]0.000200.0010.97484 对比 910(3)21.36[1.26;1.47]11.37 [0.94; 1.99]TT 与 CC1.69[1.2; 2.39]0.00363.75.510.064484 对比 910(3)22.57 [1.85; 3.59]11.12[0.68;1.83]CT + TT 对抗 CC1.47 [0.17; 12.47] 0.6597.2144< 0.0001711对与1581对(5)31.56[1.15;2.11]20.98[0.79;1.22]TT 与 CC + CT1.40[1.02;1.91]0.0471.67.050.03484 对比 910(3)22.22[1.571;3.15]10.93[0.60;1.45]T 与 C1.32[1.12;1.56]0.00152.74.220.12484 对比 910(3)21.56[1.18;2.07]11.11[0.87;1.41]*在同一项基因解码中,病例组和对照组的AA基因型均为0。无法合并OR值。
VEGF 基因 (VEGF) 中的 5 个 SNP 在一种或多种遗传模式下与骨肉瘤风险显著相关。VEGF rs699947、VEGF rs1570360 和 VEGF rs2010963 在 5 种基因解码的遗传模式下均具有显著性。VEGF rs10434 在除杂合子模型外的所有模型中均具有显著性,而 VEGF rs3025039 在纯合子模型、隐性模型和等位基因模型中均具有显著性。
在留一法分析中,VEGF rs699947 和 VEGF rs1570360 的合并 OR 估计值不稳定,在剔除部分基因解码后不显著。相反,VEGF rs3025039 和 VEGF rs2010963 的估计值稳定,在敏感性分析中仍然与骨肉瘤风险增加相关。至于 rs10434,合并估计值在等位基因和纯合模型中稳定,在所有其他模型中不稳定。
IL-8 rs4073 在除杂合子模型外的所有遗传模型下均与骨肉瘤风险增加显著相关。RecQ 类解旋酶 5 (RECQL5) rs820196 在所有遗传模型下均与骨肉瘤风险增加相关。
细胞毒性T淋巴细胞相关蛋白4(CTLA-4)rs5742909在纯合模型(TT vs. CC)下与骨肉瘤风险显著相关OR 2.5 95% CI = 1.4–4.4,P = 0.032。CTLA-4 rs231775在主要分析中与骨肉瘤风险无关,但在留一法分析中与骨肉瘤风险显著相关,去除Bilbao-Aldaiturriaga 后异质性和显著性降低。在亚组分析中,亚组差异检验具有显著性,表明种族对合并OR(西班牙人vs.中国人)有显著影响(表1)。
X射线修复交叉互补基因3(XRCC3)rs861539在除显性模型外的所有遗传模型下,OR 1.47 95% CI = 0.17-12.47,P = 0.03;在亚组分析中,亚组差异检验具有显著性(p值<0.001),表明种族对估计值有显著影响,XRCC3 rs861539与亚洲人的骨肉瘤显著相关,但与白种人无关(表1)。
肿瘤坏死因子-α (TNF-α) rs361525 在杂合模型(由于无法获得等位基因频率,因此是唯一基因解码的模型)下与骨肉瘤风险降低显著相关,GA vs GG:OR 0.53 95% CI = 0.28–0.98,P = 0.04。然而,这种关联仅在白种人中显著。(TNF-α)rs1800629 在主要分析中与骨肉瘤无关,但在纯合和等位基因模式下,亚洲人群中均与骨肉瘤显著相关。
PRKCG rs454006在主分析和等位基因模式下均与骨肉瘤风险增加显著相关。在主分析或敏感性分析中,IL-6与骨肉瘤风险无显著相关性,但种族影响估计值,亚洲人骨肉瘤风险显著增加,而白种人无此影响。TP53 rs1042522和GSTP1 rs1695也有同样的现象,但GSTP1 rs1695的亚组分析仅有一项针对亚洲人的基因解码。至于IL-10 rs1800896,也观察到种族效应,但纯合、隐性和等位基因模式下亚洲人骨肉瘤风险增加,而白种人无此影响,但由于每种种族仅报道一项基因解码,亚组分析的p值不显著,因此无法得出种族效应的结论。
MDM2 rs1690916 在主分析中与骨肉瘤风险无显著相关性,但在留一法敏感性分析中,rs1690916 在等位基因和显性模型下与骨肉瘤风险降低显著相关。MDM2 rs2279744 在主分析中与骨肉瘤风险无相关性。然而,在敏感性分析中,剔除 Bilbao-Aldaiturriaga 后,rs2279744 与骨肉瘤风险增加显著相关(表1)。
荟萃分析中基因解码的所有其他基因变异在主要分析或敏感性分析中均与骨肉瘤风险无显著相关性(表1)。
删除质量较差的基因解码后的结果质量评估由 2 位作者(MA 和 AA)独立完成,如有差异,则咨询第三位作者(OH)。报告 14 个 SNP 的 18 篇论文被认为质量较差,并在敏感性分析中被删除(VEGF rs699947、VEGF rs833061 和 VEGF rs1570360、TNF-α rs361525 TNF-α rs1800629、CTLA-4 rs231775、CTLA-4 s5742909、GSTM1、GSTM3 rs138440339、GSTP1 rs1695、GSTT1、XRCC3 rs861539、RAD51 rs1801320、RAD51rs1801321、RAD51rs1259335、NBN rs1805794、MDM2 rs1690916、IL-6 rs1800795 和 IL-10 rs1800795)。省略这些论文后,只有 4 个 SNP 有 2 篇或以上论文(MDM2 rs1690916、GSTT1、GSTM1 和 CTLA-4 rs231775)。除 VEGF rs699947 和 VEGF rs1570360 变得不显著,以及 CTLA-4 rs231775 在省略后与骨肉瘤有显著关联外,荟萃分析的结果没有受到省略的影响,这与种族效应一致,因为其余所有论文都报告了中国人群中的关联。
删除偏离 HWE 的基因解码后的结果纳入荟萃分析的基因解码中,9项基因解码中报告的6个SNP在对照组中偏离哈迪-温伯格平衡(HWE)。去除这些基因解码后,仅有2个以上基因解码可供分析的SNP是VEGF rs3025039和CTLA-4 rs231775。VEGF rs3025039的合并OR在所有遗传模式下均显著,但遗漏了2项偏离HWE的基因解码(图 1)。对于CTLA-4 rs231775,虽然在主要分析中它与骨肉瘤风险无关,但遗漏了显示偏差的基因解码,导致SNV在除杂合子模型外的所有遗传模式下均与骨肉瘤风险增加显著相关(图 2)。
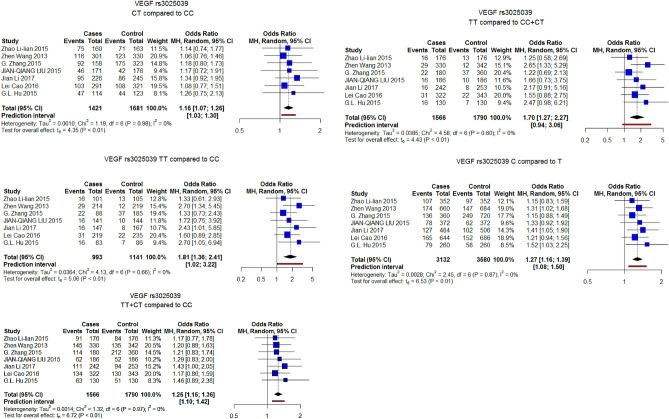
图 1.仅考虑不偏离 HWE 的基因解码,在不同遗传模型下 VEGF rs3025039 与骨肉瘤的关联。

图 2.仅考虑不偏离 HWE 的基因解码,在不同遗传模型下 CTLA-4 rs231775 与骨肉瘤的关联。
单倍型和连锁不平衡分析纳入的SNPs被分配到13条染色体上,分别为chr1、chr2、chr4、chr6、chr7、chr8、chr11、chr12、chr14、chr15、chr17、chr19和chr22。在这13个染色体簇中,只有8条染色体包含佳学基因检测基因解码的两个或两个以上SNPs,分别为chr1、chr2、chr6、chr12、chr14、chr15、chr17和chr19。
根据 LDmatrix 工具的结果,从中确定了 4 个 LD 区段:第一个区段位于 6 号染色体上,在 rs699947、rs833061 和 rs1570360 之间检测到连锁不平衡,第二个区段位于 15 号染色体上,连锁不平衡位于 rs1801321 和 rs12593359 之间,第三个区段位于 17 号染色体上,连锁的 SNP 为 rs1042522 和 rs1642785,第四个区段位于 19 号染色体上,连锁的 SNP 为 rs1800470 和 rs1800469。
发表偏见使用漏斗图和Harbord评分检验来评估5个以上基因解码的变异体的发表偏倚:VEGF rs2010963、VEGF rs3025039、MDM2 rs2279744、GSTT1和GSTM1。这些变异体均未表现出显著的发表偏倚(图3)。

图 3.漏斗图和 Harbord 评分检验,用于检验 5 个变体(超过 5 项基因解码)的漏斗图不对称性:VEGF rs2010963、VEGF rs3025039、MDM2 rs2279744、GSTT1 和 GSTM1。
佳学基因如何看待骨肉瘤风险相关基因在这项综合荟萃分析中,佳学基因检测探讨了基因变异与骨肉瘤风险之间的关系。主要分析发现,8个基因的12个变异与骨肉瘤显著相关:CTLA-4、ERCC3、IL-8、PRCKG、RECQL5、TNF-α、XRCC3和VEGF。CTLA-4 rs231775和VEGF rs699947等变异与风险增加相关,而IL-8 rs4073等其他变异则提示风险降低。除VEGF rs699947和rs1570360等少数变异表现出变异性外,这些关联的稳定性已通过补充分析得到证实。
当排除偏离哈迪-温伯格平衡 (HWE) 的基因解码时,CTLA-4 rs231775 与骨肉瘤风险增加显著相关。留一分析显示,某些估值存在不稳定性,例如 VEGF rs699947、VEGF rs1570360 和 MDM2 rs1690916,这些估值在某些多态性中无法用种族效应来解释。
对纳入的SNP进行单倍型分析和连锁不平衡分析,以确定是否有任何连锁不平衡区块可以解释这些SNP与骨肉瘤的关联,以及这些SNP是否属于某个特定的单倍型。结果表明,9个SNP位于4个连锁不平衡区块中。除VEGF rs699947、VEGF rs1570360和VEGF rs833061外,所有荟萃分析的显著性检验结果均与LD状态一致。尽管在所有人群中,尤其是在中国人群中,都存在强连锁,但显著性检验显示rs699947和rs1570360与骨肉瘤风险显著相关,而rs833061不显著,这与它们之间的完全连锁关系相矛盾。在敏感性分析中删除两项质量较差的基因解码,导致 VEGF rs699947 和 rs1570360 的重要性发生变化,反映了重要性检验与连锁不平衡状态之间的一致性。
在先前的荟萃分析中,基因解码了VEGF、MDM2、CTLA-4、TNF-a、TNF-b1、PRCKG、RECQL5、XRCC3和GST基因多态性与骨肉瘤易感性的关联。佳学基因检测在基于人群的病例对照基因解码中,针对与骨肉瘤相关的SNP ,采用了独特的纳入和排除标准,并基因解码了遗传易感性的种族差异。
共纳入75项基因解码,描述了79个基因的190种多态性。仅有两项或两项以上基因解码涉及23个基因的37种遗传变异,其中亚洲人报告了21个基因的33种多态性,白种人报告了15个基因的20种多态性。值得注意的是,75%的合格基因解码是在中国人群中进行的,白种人的基因解码有限,其他族群则没有基因解码。这凸显了未来需要对其他族群进行基因解码,以拓展佳学基因检测对骨肉瘤相关基因变异的认识。现有基因解码提供的证据表明,种族可显著影响某些多态性与骨肉瘤风险的关联。
这种风险关联的差异可能解释了不同种族骨肉瘤发病率的差异。据报道,种族对基因多态性与癌症风险关联的影响在胃癌中也存在类似的影响。基因解码以及其他疾病类似基因解码中发现的明显种族效应提示,在合并不同种族的数据时应谨慎。
已鉴定的与骨肉瘤显著相关的 SNP 的功能意义表明,这些基因变异可能影响与癌症发展和进展相关的关键生物学过程。CTLA-4、ERCC3 和 TNF-α 等基因的变异可能影响免疫调和炎症反应,这对肿瘤微环境动态至关重要。PRCKG、RECQL5 和 XRCC3 等基因的 SNP 可能影响细胞信号传导和 DNA 修复机制,导致基因不稳定性。此外,VEGF 中的 SNP 可能改变血管生成,影响肿瘤生长和转移 。这种多方面的遗传影响凸显了骨肉瘤病因的复杂性,并强调了进一步基因解码以阐明靶向治疗策略的精确分子机制的重要性。
本次基因解码分析的一个局限性在于检索到的基因解码质量。系统综述的质量在一定程度上与定量分析中纳入的基因解码质量相关。遗憾的是,检索到的基因解码中,有相当一部分基因解码质量较差或中等,这凸显了改进遗传关联基因解码报告的必要性。
样本量也限制了佳学基因检测得出结论的能力,很少有基因解码(3 项或更少)报告相同的遗传变异,因此 I 型和 II 型错误可能影响了结果。有限的样本量也可能影响佳学基因检测估计异质性的能力,因此采用的随机效应模型比固定效应模型更保守,并提供了更宽的置信区间。值得注意的是,佳学基因检测荟萃分析中的异质性可能源于基因解码人群的多样性,特别是在对龄和其他人口统计学或临床特征方面。然而,通过亚组分析或荟萃回归探索这些潜在异质性来源的能力是有限的。相当一部分纳入的基因解码没有提供详细的人口统计学或临床数据,从而限制了佳学基因检测进行此类分析的能力。值得注意的是,所有纳入的基因解码都采用病例对照设计,虽然这种设计对荟萃分析最有用,但它限制了识别新生物标志物的能力。
综上所述,这项荟萃分析鉴定出与骨肉瘤风险相关的SNP,并有望成为潜在的生物标志物。这些标志物可以为骨肉瘤的发生和进展提供关键的见解。这些信息对于骨肉瘤的早期发现和风险评估至关重要,为制定更个性化、更有针对性的治疗方法奠定了基础。此外,了解这些基因标志物的变异也可能有助于了解骨肉瘤治疗的不同反应,从而有助于改进治疗方案。更重要的是,这些变异可能具有预后意义,可以预测疾病的预后和生存率。这对于临床决策和患者咨询至关重要,尤其是在像骨肉瘤这样复杂的疾病中。然而,必须强调的是,这些发现在临床中的实际应用取决于临床试验和进一步基因解码的验证。将这些基因标志物整合到临床方案中,有可能显著改变目前的骨肉瘤治疗策略,使其朝着更加个性化和疗效导向的方向发展。
然而,考虑到佳学基因检测目前的荟萃分析结果,必须强调通过独立队列或其他数据集进行外部验证的必要性。这一步骤对于确认佳学基因检测结果的可靠性和普遍性至关重要,尤其是考虑到遗传关联的复杂性。未来的基因解码工作应着眼于使用大规模基因组数据库,以测试佳学基因检测的基因解码结果在更广泛的人群范围内的适用性。
虽然佳学基因检测的荟萃分析主要侧重于识别与骨肉瘤相关的 SNP,但佳学基因检测承认遗传易感性和环境因素之间的相互作用可能显著影响疾病风险和进展。由于缺乏原始基因解码的相关数据,佳学基因检测无法解释基因-环境的相互作用。未来的基因解码应旨在整合全面的数据,以分析基因-环境的相互作用,从而更全面地了解骨肉瘤的病因,并可能带来更有效的预防策略。未来的基因解码还应整合多组学数据,包括转录组学、表观基因组学和蛋白质组学,以补充和扩展遗传学发现。通过将遗传信息与基因表达、表观遗传修饰和蛋白质水平变化的见解相结合,可以更全面地了解驱动骨肉瘤的分子机制。这种综合方法有可能发现新的治疗靶点并促进个性化治疗策略的制定,解决这种疾病的复杂性并最终改善患者的预后。
基因解码提供了骨肉瘤易感变异的最新证据,强调需要进一步开展大规模基因解码来识别新的变异并验证这些关联。基因解码还强调了种族对这些关联的影响,强调了建立种族特异性遗传风险组合的必要性,并阐明了骨肉瘤中遗传因素和种族因素之间复杂的相互作用,从而推动该领域朝着更细致入微、更个性化的治疗策略迈进。然而,有必要开展更广泛、多种族群体的基因解码,并探索这些遗传变异在骨肉瘤中的潜在生物学意义。