卵巢交界性肿瘤(BOTS)是一类罕见的卵巢肿瘤,其生物学行为介于良性的囊腺瘤与低级别卵巢癌(lgOvCa)之间。在分子层面上,BOTS 与 低级别卵巢癌(lgOvCa)表现出一定相似性。然而,与基因解码相对充分的高级别卵巢癌(hgOvCa)相比,BOTS 和 低级别卵巢癌(lgOvCa)的分子特征尚不明确。
本基因解码采用新一代测序(NGS)技术,分析伴或不伴 BRAF V600E 突变的 BOTS、低级别卵巢癌(lgOvCa)和高级别卵巢癌(hgOvCa)中关键抑癌基因及致癌基因的遗传变异情况。随后,通过桑格测序验证所选基因多态性的存在,并利用蛋白质印迹法(Western blot)评估这些变异对相应蛋白表达的影响。
基因解码结果揭示了不同卵巢肿瘤类型在多态性模式上的差异,提示它们可能涉及不同的信号通路。在未携带 BRAF V600E 突变的 BOTS 中,KRAS 突变似乎具有重要作用;而在 低级别卵巢癌(lgOvCa)中,KRAS 和 NRAS 突变则更为常见。相较之下,这些突变在高级别卵巢癌(hgOvCa)中的出现频率较低。
此外,通过多变量回归分析,佳学基因检测识别出 BOTS 中的 PARP1 以及高级别卵巢癌(hgOvCa)中的 FANCI、BRCA2、TSC2 和 FANCF 等潜在生物标志物。值得一提的是,在某些关键基因(如 FANCI、FANCD2、FANCF、TSC2 以及 BRCA1/2、TP53)中观察到的遗传状态,对于理解疾病进展和预后具有重要意义。
本基因解码为不同侵袭性卵巢肿瘤间的分子异质性提供了新视角,并提出了若干可能作为预后评估或治疗反应预测指标的分子标志物。
关键词:卵巢癌、交界性卵巢肿瘤、多态性、NGS、蛋白质印迹、TP53、RAS、BRAF、BRCA1/2
一、基因解码背景卵巢癌(OvCa)是一种临床上常见且高度异质的女性恶性肿瘤,全球范围内其预后较差、死亡率高。根据病理分型,卵巢癌主要分为高级别(hgOvCa)和低级别(lgOvCa)两类。hgOvCa 是最常见的类型,通常表现为高度基因组不稳定、染色体结构异常以及多个关键肿瘤抑制基因(如 TP53、BRCA1、BRCA2)的高频突变。
相比之下,低级别卵巢癌(lgOvCa)较为罕见,通常在较年轻的患者中发现,虽然其对化疗反应较差,但生存期通常更长。低级别卵巢癌(lgOvCa)中 TP53 和 BRCA1/2 的突变频率明显低于 hgOvCa,尤其在其浆液性亚型中,与 BOTS 的分子谱存在重叠。
BOTS 是一类生物学行为介于良性和恶性之间的肿瘤,大多见于育龄女性,占所有上皮性卵巢肿瘤的约 15%。这些肿瘤多数在 FIGO 早期被诊断,整体预后良好。然而,由于缺乏特异性的影像学表现,术前诊断存在困难。此外,即便进行完全切除,约有 20% 的 BOTS 仍可能复发,其中近 30% 的复发病例最终发展为浸润性卵巢癌。
在分子水平上,BOTS 与高级别卵巢癌(hgOvCa)存在显著差异。BOTS 中较少出现 TP53 和 BRCA1/2 的突变,最常见的是 BRAF 和 KRAS 突变,特别是在浆液性亚型中,这些变异有时也可见于 lgOvCa,而在高级别卵巢癌(hgOvCa)中则较为罕见。此外,一些基因解码亦关注了如 PIK3CA、EGFR、CTNNB1、RAD51C、PALB2、CHEK2 和 PTEN 等基因在 BOTS 中的突变频率。然而,关于 BOTS 中抑癌基因与致癌基因的多态性信息仍然有限。
相较而言,hgOvCa 的遗传特征基因解码更为深入,但部分分子标志物的临床意义仍存在争议。因此,深入探讨 BOTS、低级别卵巢癌(lgOvCa)和高级别卵巢癌(hgOvCa)的分子图谱,具有重要的临床和基因解码价值。
二、基因解码目的与方法本基因解码使用两套新一代测序(NGS)基因面板,对携带或不携带 BRAF V600E 突变的 BOTS(分别称为 BOT 和 BOT.V600E),以及 低级别卵巢癌(lgOvCa)和高级别卵巢癌(hgOvCa)样本中 76 个相关基因的多态性状态进行分析:
第一套基因面板: 涵盖 41 个与遗传性卵巢癌密切相关的致癌基因和抑癌基因,以及 CRNDE、IRX5 和 CEBPA;
第二套基因面板: 包括 37 个在散发性人类癌症中常见的突变热点,大多数不包括在第一套面板中。
基因解码流程包括:
基因多态性的单变量与多变量统计分析;
关键位点的桑格测序验证;
蛋白质印迹实验,验证基因变异与蛋白表达间的关联性。
本基因解码旨在揭示不同类型卵巢肿瘤的基因多态性特征,为后续生物标志物的开发和疾病机制的解析奠定基础。
2. 结果2.1 不同肿瘤组基因多态性的分布经过NGS和生物信息学分析后,佳学基因检测获得了一系列对相应蛋白质结构和功能具有高度或中度影响的基因变异。对这些具有不同影响的基因变异进行了组合分析或单独分析,以确定具有不同影响的变异对卵巢肿瘤结果的影响是一致还是不一致。此外,还有两个问题需要澄清。首先,在44个基因的面板中,一个额外的致癌基因KCNMB3 被富集,该基因是非预期的基因解码对象。这可能是因为它的基因座与PIK3CA基因的基因座部分重叠,而 PIK3CA 基因最初包含在面板中,由相反的 DNA 链编码。同样,在热点面板中,一个额外的基因FBXW7-AS1被富集,它是热点面板中存在的FBXW7基因 [ 17 ]的反义转录本。其次,佳学基因检测团队在另一篇论文中描述了CRNDE基因(在 44 基因组中添加)的多态性,因此本文不再赘述。
当在整个卵巢肿瘤组中使用 44 个基因面板时,佳学基因检测发现了 85 种独特的、以前未描述过的变异(71 个新的 SNP 和 14 个新的非 SNP)。。图 1 A、B(具有高或中等影响 (A) 或仅为高影响 (B) 的变异)。在图 1 C-F 和图 S1C、E中,显示了相关的箱线图,分别描绘了不同卵巢肿瘤组中 SNP 和非 SNP 变异的总体频率。这些箱线图还补充了详细的统计测试。此外,图 S2A - F 还显示了每个肿瘤组每个基因的 SNP 或非 SNP 变异的平均计数。当综合考虑所有变异时(图 1 A),最常改变的基因(至少一个组中改变的样本比例 > 0.5)是BRCA1、BRCA2、FANCA、SEM1和TP53 。但是,如果仅考虑高影响变异(图 1 B),则遗传变异频率最高(> 0.3)的是BRCA1和TP53基因,以及高级别卵巢癌(hgOvCa)组。相比之下,与所有剩余肿瘤组相比,在没有BRAF V600E 突变的BOT 中,对蛋白质结构/功能具有高/中等影响(图 1 C)或仅有中等影响(图 S1C )的 SNP 数量明显更高。此外,相同的分析显示,BOT.V600E 肿瘤的特征是 SNP 数量明显低于 hgOvCa。值得注意的是,在高级别卵巢癌(hgOvCa)中,与所有其他肿瘤组相比,高影响基因变异(SNP 或非 SNP)的数量显著升高,除了在 低级别卵巢癌(lgOvCa)与高级别卵巢癌(hgOvCa)比较中影响较大的 SNP(图 1 D-F)。值得注意的是,对蛋白质结构/功能仅有中等影响的非 SNP 数量并未显著区分任何卵巢肿瘤组(图 S1E),这可能是因为这些变异的频率较低(仅在三个基因ATRX、CHEK1和PTEN中发现了六种此类变异,仅在 9 个高级别卵巢癌(hgOvCa)肿瘤中发现。

图 1.SNP 和非 SNP 变异——44 个基因面板。(A、B)每个肿瘤组中每个基因的所有变异的累积频率((A)—具有高或中等影响的变异,(B)—仅具有高影响的变异)。(C – F)箱线图显示所分析的肿瘤组之间 SNP((C)高或中等影响,(D)仅高影响)和非 SNP((E)高或中等影响,(F)仅高影响)的遗传变异数量差异。每个箱线图还补充了 Kruskal-Wallis 秩和检验(显示所分析的变异集之间是否存在统计学上显着差异)和带有连续性校正的 Wilcoxon 秩和检验(事后检验用来确定哪些肿瘤组彼此不同)。NS:不显着。组大小:BOT:n = 53; BOT.V600E:n = 23; lgOvCa:n = 10; hgOvCa:n = 139。
对于热点面板,佳学基因检测发现了 82 种独特的、以前未描述过的遗传变异(75 个新的 SNP 和 7 个新的非 SNP)。它们的列表可以在Supplement-variants.xlsx中找到。图 2 A、B(具有高或中等影响(A)或仅为高影响(B)的变异)和图 S1B(仅为中等影响的变异)显示了每组肿瘤中所有基因中检测到的所有变异(SNP 和非 SNP 合并)的累积频率。在图 2 C-F 和图 S1D、F中,显示了相关的箱线图,分别描绘了不同卵巢肿瘤组中 SNP 和非 SNP 变异的总体频率。这些箱线图还补充了详细的统计测试。此外,图 S2G -L还显示了每个肿瘤组每个基因的 SNP 或非 SNP 改变的平均计数。当同时考虑高影响变异和中度变异,以及 SNP 和非 SNP 时(图 2 A),最常改变的基因(至少一个组中改变的样本比例 > 0.5)是PTCH1(在所有肿瘤组中均发生改变)和TP53(主要在高级别卵巢癌(hgOvCa)中发生改变)。有趣的是,在热点面板中,BRCA1基因中的遗传变异的识别频率低于在 44 基因面板中的频率。然而,如果仅考虑高影响变异,则使用两个面板检测到的BRCA1和TP53的突变谱相似,揭示了在 OvCa 中这些基因的遗传变异频率很高,尤其是在高级别卵巢癌(hgOvCa)中(图 1 B 和图 2 B)。值得注意的是,在热点面板中,佳学基因检测还在一个lgOvCa样本中检测到了TP53基因的两个变异(chr17:g.7674921C>A,p.Glu204Ter和chr17:g.7676218C>A,p.Glu51Ter)。这两个SNP在44个基因面板中均未发现,可能是因为它们的频率较低,分别为11%和14%。需要在此提及的是,在佳学基因检测的生物信息学工作流程中,所有频率低于10%的序列变异都被过滤掉,以消除由DNA聚合酶错误引起的变异,以及那些过于罕见以至于既无法产生明显的临床效果也无法通过桑格测序成功验证的变异。
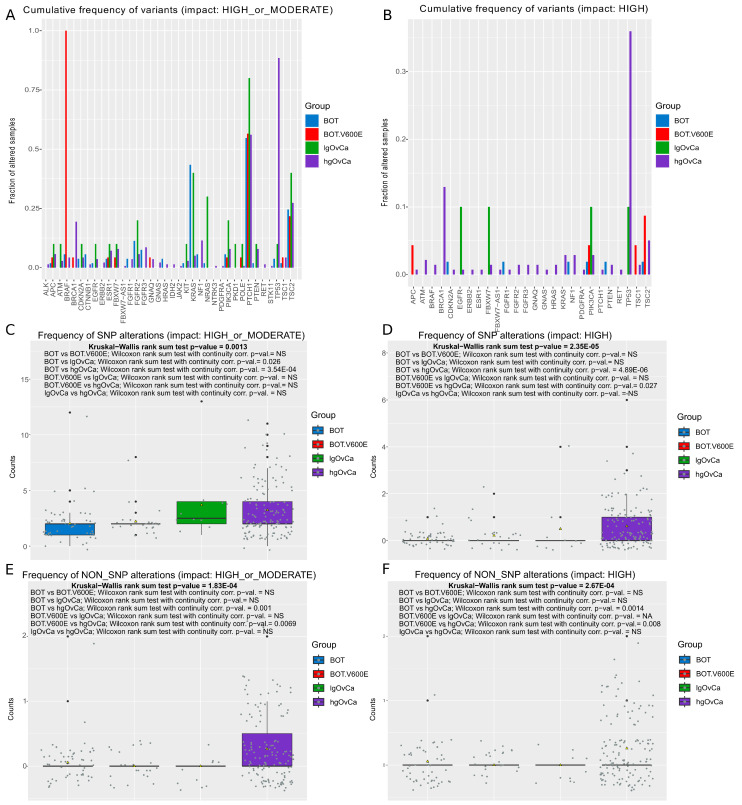
当仅考虑 SNP 时,热点面板的结果与 44 基因面板的结果在 BOT 组中非高影响 SNP 的频率方面存在重要差异。在热点面板中,BOT 中此类 SNP 的数量明显低于两个 OvCa 组(图 2 C 和图 S1D)。相比之下,在 44 基因面板中,BOT 中的非高影响 SNP 比所有其他肿瘤组都丰富得多(图 1 C 和图 S1C)。然而,当考虑高影响 SNP 或所有非 SNP 时,这种分歧消失了(图 1 D-F 和图 2 D-F),表明在两个面板中高级别卵巢癌(hgOvCa)的基因改变频率均高于 BOTS。
将每个样本每个基因的所有 SNP 和非 SNP 变异相加并二值化(至少存在一个变异 vs. 无变异)。随后对该数据集进行的统计分析(如表 1所示)显示,无论基因组合和变异影响如何, TP53都是低侵袭性肿瘤(BOT、BOT.V600E 和 lgOvCa)与 hgOvCa(后者突变频率更高)之间最具区分性的基因。该规则的唯一例外是热点组合中 低级别卵巢癌(lgOvCa)与高级别卵巢癌(hgOvCa)对比中存在高影响变异,未观察到统计学显著性。
表 1.通过两个基因组识别出对卵巢肿瘤组具有高度或中等影响的遗传变异,可显著区分卵巢肿瘤组。44个基因面板影响高或中等组比较和p值基因BOT 与 BOT.V600EBOT 与 lgOvCaBOT 与 hgOvCaBOT.V600E 与 lgOvCaBOT.V600E 与 hgOvCa低级别卵巢癌(lgOvCa)与 hgOvCaTP535.67 × 10 −31 (↑hgOvCa)1.23 × 10 −18 (↑hgOvCa)1.8 × 10 −9 (↑hgOvCa)FANCB9.71 × 10 −3(↑BOT)SEM12.51 × 10 −2(↑底部)1.01 × 10 −2(↑底部)范卡2.61 × 10 −2(↑BOT)FANCD24.97 × 10 −2 (↑hgOvCa)1.52 × 10 −2 (↑hgOvCa)BRCA21.47 × 10 −2 (↑底部)CHEK21.04 × 10 −2(↑底部)MUTYH1.44 × 10 −2 (↑底部)RAD502.83 × 10 −2(↑底部)影响中等组比较和p值基因BOT 与 BOT.V600EBOT 与 lgOvCaBOT 与 hgOvCaBOT.V600E 与 lgOvCaBOT.V600E 与 hgOvCa低级别卵巢癌(lgOvCa)与 hgOvCaTP533.48 × 10 −14 (↑hgOvCa)6.97 × 10 −9 (↑hgOvCa)1.64 × 10 −4 (↑hgOvCa)BRCA12.76 × 10 −2(↑底部)FANCB9.71 × 10 −3(↑BOT)SEM12.51 × 10 −2(↑底部)1.01 × 10 −2(↑底部)MUTYH3.8 × 10 −2 (↑底部)BRCA23.83 × 10 −3(↑底部)CHEK25.94 × 10 −3(↑BOT)FANCA2.61 × 10 −2(↑BOT)FANCD24.97 × 10 −2 (↑hgOvCa)1.52 × 10 −2 (↑hgOvCa)RAD502.83 × 10 −2(↑底部)PALB24.31 × 10 −2(↑底部)ATM3.62 × 10 −2 (↑lgOvCa)高影响组比较和p值基因BOT 与 BOT.V600EBOT 与 lgOvCaBOT 与 hgOvCaBOT.V600E 与 lgOvCaBOT.V600E 与 hgOvCa低级别卵巢癌(lgOvCa)与 hgOvCaTP531.25 × 10 −8 (↑hgOvCa)1.47 × 10 −4 (↑hgOvCa)3.08 × 10 −2 (↑hgOvCa)BRCA11.25 × 10 −7 (↑hgOvCa)6.01 × 10 −4 (↑hgOvCa)3.4 × 10 −2 (↑hgOvCa)热点面板影响高或中等组比较和p值基因BOT 与 BOT.V600EBOT 与 lgOvCaBOT 与 hgOvCaBOT.V600E 与 lgOvCaBOT.V600E 与 hgOvCa低级别卵巢癌(lgOvCa)与 hgOvCaTP531.01 × 10 −29 (↑hgOvCa)7.62 × 10 −18 (↑hgOvCa)2.35 × 10 −7 (↑hgOvCa)BRAF1.52×10−16 ( ↑BOT.V600E)1.08×10−8 ( ↑BOT.V600E)1.08× 10−23(↑BOT.V600E)NRAS1.1 × 10 −2 (↑lgOvCa)2.2 × 10 −2 (↑lgOvCa)2.22 × 10 −4 (↑lgOvCa)BRCA11.08 × 10 −4 (↑hgOvCa)FBXW73.67 × 10 −2 (↑hgOvCa)KRAS6.44 × 10 −5(↑底部)2.58 × 10 −10 (↑底部)5.13 × 10 −3 (↑lgOvCa)2.77 × 10 −3 (↑lgOvCa)影响中等组比较和p值基因BOT 与 BOT.V600EBOT 与 lgOvCaBOT 与 hgOvCaBOT.V600E 与 lgOvCaBOT.V600E 与 hgOvCa低级别卵巢癌(lgOvCa)与 hgOvCaTP532.27 × 10 −15 (↑hgOvCa)1.66 × 10 −9 (↑hgOvCa)8.26 × 10 −5 (↑hgOvCa)BRAF1.52×10−16 ( ↑BOT.V600E)1.08×10−8 ( ↑BOT.V600E)1.08× 10−24(↑BOT.V600E)NRAS1.1 × 10 −2 (↑lgOvCa)2.2 × 10 −2 (↑lgOvCa)2.22 × 10 −4 (↑lgOvCa)KRAS6.44 × 10 −5(↑底部)1.41 × 10 −11 (↑BOT)5.13 × 10 −3 (↑lgOvCa)1.11 × 10 −3 (↑lgOvCa)高冲击力组比较和p值基因BOT 与 BOT.V600EBOT 与 lgOvCaBOT 与 hgOvCaBOT.V600E 与 lgOvCaBOT.V600E 与 hgOvCa低级别卵巢癌(lgOvCa)与 hgOvCaTP535.84 × 10 −9 (↑hgOvCa)1.35 × 10 −4 (↑hgOvCa)BRCA13.98 × 10 −3 (↑hgOvCa)包含适用检验(卡方或 Fisher 精确概率法)的p值,后跟箭头和特定基因发生更频繁改变的组名(均写在括号中)。如果缺乏统计学显著性,则相应的单元格为空。
这里值得一提的另外两个基因是BRCA1和BRCA2,因为在本基因解码中,它们的突变谱似乎不仅取决于所使用的基因组,还取决于基因变异对这些基因编码的蛋白质的结构和功能的影响。在 44 基因组中,如果考虑中等影响变异,则上述两个基因在 BOT 中的变异频率高于 hgOvCa。如果包括BRCA2基因中影响较大的变异,这种规律仍然适用。相比之下,只有影响较大的BRCA1变异在高级别卵巢癌(hgOvCa)中出现的频率远高于所有其他卵巢肿瘤组(表 1)。在热点组中,不包括BRCA2基因,而无论仅考虑影响较大的变异还是考虑所有基因变异,hgOvCa中的BRCA1多态性数量都明显高于 BOT。
在本基因解码中,KRAS基因的高强度或中等影响变异最能将 BOT 与除 低级别卵巢癌(lgOvCa)之外的所有其他肿瘤组区分开来。在另外两个参与泛素化的基因FANCB和SEM1中,中等影响变异在 BOT 中的出现频率明显高于在 OvCa ( SEM1 ) 或高级别卵巢癌(hgOvCa)( FANCB ) 中出现的频率。值得注意的是,这两个基因的变异并不能将 BOT 与 BOT.V600E 区分开来。此外,在本基因解码的两个小组基因解码的 76 个不同基因中,BRAF是唯一一个在 BOT.V600E 肿瘤中突变频率高于所有其他组的基因。
与 BOT.V600E 和高级别卵巢癌(hgOvCa)相比, KRAS基因变异不仅在 BOT 中频繁发生,在 低级别卵巢癌(lgOvCa)中也同样频繁发生。除KRAS外,另外两个基因ATM和NRAS的变异在 低级别卵巢癌(lgOvCa)中也较为常见。在 低级别卵巢癌(lgOvCa)中,ATM 的变异频率高于在 BOT.V600E 组中,但这种规律性仅限于中等影响的变异。至于NRAS,与其余三个肿瘤组相比,该基因的中等影响变异在 低级别卵巢癌(lgOvCa)中较为常见。
为了验证所选基因的多态性,佳学基因检测采用了梯度PCR结合桑格测序的方法。通过这项技术,佳学基因检测成功验证了TP53基因中一个先前发现的变异(chr17:g.7670658_7670659insA,p.Lys351Ter)以及七个新的变异(SNP和非SNP),这些变异对蛋白质的结构/功能具有中等或高度影响。验证结果及每个分析多态性的详细描述如图S3所示。
基因解码如何提高基因检测和临床治疗的有效性本基因解码旨在分析不同侵袭性卵巢肿瘤中关键抑癌基因和致癌基因的遗传变异。佳学基因检测不仅评估了这些基因在大规模、特征明确的卵巢癌(OvCa)和卵巢癌旁组织(BOTS)患者队列中的多态性状态,还发现了这两组肿瘤的预测和/或预后标志物,并分析了特定多态性对相应蛋白质表达的影响。
出乎意料的是,佳学基因检测对 44 个基因面板获得的 NGS 结果显示,与 BOT.V600E、低级别卵巢癌(lgOvCa)或高级别卵巢癌(hgOvCa)相比,BOT 中对相应蛋白质的结构和/或功能具有高度或中度或仅有中度影响的 SNP 数量更高。相反,当仅分析选定基因中的热点时,BOT 中具有这些影响的 SNP 变体的频率明显低于两个 OvCa 组。这种明显的差异可以通过本基因解码中调查的两个面板包含不同的基因集来解释。如本基因解码所证明的,与其他肿瘤组(FANCB、SEM1、FANCA、BRCA2、CHEK2、MUTYH、RAD50)相比,BOT 中 44 个基因面板中突变更频繁的基因列表比热点面板(仅KRAS )中类似改变的基因长得多。此外,热点组的设计初衷是基因解码众所周知的基因变异。相比之下,44基因组覆盖的基因组区域大约大10倍,能够检测出通常在诊断方法中被忽略的罕见遗传变异。然而,当仅考虑对蛋白质功能和/或结构影响较大的多态性时,两个组中鉴定出的遗传变异数量在hgOvCa中最高,从而支持了关于卵巢癌的普遍认识。
TP53的突变状态可被认为是区分 hgOvCa( TP53频繁突变)与 BOTS(无突变或非常罕见突变)和 lgOvCa(相对罕见突变)的最佳标记之一。与这些报告一致,TP53 是本基因解码中最常见的变异基因之一,主要在高级别卵巢癌(hgOvCa)中。相比之下,除了在 低级别卵巢癌(lgOvCa)样本中发现了两个覆盖率较低且影响较大的 SNP 外,佳学基因检测在 低级别卵巢癌(lgOvCa)病例中未发现TP53变异。有趣的是,这些 SNP 仅在热点面板中检测到,利用了一种新颖的 NGS 杂交捕获技术(称为引物延伸靶向富集,KAPA HyperPETE,Roche),比 44 基因面板中使用的较旧的基于杂交的捕获方法(KAPA HyperCap,Roche)提供了更好的测序覆盖均匀性。至于 BOTS,本基因解码发现的TP53中仅有的两个错义变体是在两个粘液亚型的 BOT 样本中观察到的。鉴于 Kang 等人报道 19.4% 的粘液 BOTS 中存在 TP53 突变,这与较高的复发风险相关,该结果也与当前的知识状态相符。佳学基因检测的两名TP53突变携带 BOT 患者中始终有一名进展为 OvCa。值得注意的是,本文佳学基因检测还通过观察高影响非SNP变异样本中TP53的缺失以及含有TP53错义SNP的肿瘤中TP53的积累,在蛋白质水平上验证了TP53的NGS结果。这些结果与佳学基因检测之前对TP53表达的免疫组织化学评估结果一致。
根据文献,除了TP53的遗传畸变外, BRCA1/2的突变在高级别卵巢癌(hgOvCa)中也很常见,而在 BOTS 和 低级别卵巢癌(lgOvCa)中很少见。佳学基因检测使用 44 个基因面板获得的结果似乎与文献并不完全一致,因为佳学基因检测在许多非高级别卵巢肿瘤中发现BRCA1/2基因变异。然而,需要强调的是,除了单个 BOT 中的一个 SNP 外,这些只是中等影响的变异。与高级别卵巢癌(hgOvCa)相比,这些变异解释了BOT 中BRCA1/BRCA2中发现的遗传变异数量显著增加。然而,当只考虑高影响变异时,与所有其余肿瘤组相比, BRCA1(而不是BRCA2)在高级别卵巢癌(hgOvCa)中的改变频率如预期的那样更高。相比之下,在热点组(仅集中于已确定的变异,而忽略了大多数基因解码不足的基因变异)中,BOT 或 低级别卵巢癌(lgOvCa)中未发现具有高/中等影响的BRCA1多态性,而在一个 BOT.V600E 样本中仅存在一个中等影响的变异。因此,当仅考虑BRCA1基因中常见的热点时,佳学基因检测的统计工作流程证实了与 BOT 相比,hgOvCa 中该基因序列变异普遍占优势这一事实。尽管如此,根据最近一项针对大型队列(包含 1333 名 OvCa 患者和 152 名 BOTS 患者)进行的 NGS 基因解码,高级别卵巢癌(hgOvCa)和 BOTS 中BRCA1/2突变的患病率相似(分别为 30.9% 和 28.9%)。因此,本文似乎证实了佳学基因检测利用 44 个基因面板得出的结论,即如果应用高通量测序技术(不仅限于已知的热点),则可以在 BOTS 中检测到BRCA1中的大量遗传变异。至于BRCA2 ,与BRCA1类似,与高级别卵巢癌(hgOvCa)相比,该基因的中等影响变异在 BOT 中占主导地位。相反,佳学基因检测发现在所基因解码的卵巢肿瘤组之间,高强度BRCA2多态性的频率没有差异。尽管如此,在本基因解码中, BRCA2成为高级别卵巢癌(hgOvCa)中有希望的、有利的预测和预后标志物。BRCA2序列变异的存在改善了患者的 OS、CR 和 PS,尤其是在没有 TP53 积累的肿瘤中。虽然考虑到该基因的肿瘤抑制能力,这种结果可能看起来很奇怪,但早些时候在小细胞肺癌中也观察到了类似的现象,其中引用的基因解码的作者报告了BRCA2突变的发生与肿瘤对化疗的更高敏感性之间的联系。与这些结果一致,体外获得的数据也为 BRCA 缺陷肿瘤对铂类药物的反应更好提供了强有力的证据,体外基因解码进一步证实了这一点,其中BRCA突变携带者在接受铂类药物治疗后表现出更好的生存期和更长的无病间隔。由于 BRCA1/BRCA2 蛋白负责修复双链 DNA 断裂 (DSB),BRCA2中致病变异的存在会导致其蛋白产物活性受损,从而增加肿瘤细胞中发生 DSB 的风险。如果这样的细胞表达功能性 TP53(未观察到 TP53 积累),则会诱导细胞凋亡,从而改善铂类治疗的效果,如本文所示。
至于低侵袭性卵巢肿瘤的基因变异特征,与高级别卵巢癌(hgOvCa)相比,BOTS 和 低级别卵巢癌(lgOvCa)中多态性数量最多的基因是KRAS、BRAF和NRAS,这与科学文献 。鉴于携带BRAF V600E 变异的 BOTS 患者比未携带该变异的患者年轻得多,因此本文对这两组肿瘤分别进行了分析。有趣的是, BOT 和 低级别卵巢癌(lgOvCa)中KRAS的突变频率高于 BOT.V600E 或 hgOvCa,而 低级别卵巢癌(lgOvCa)和 BOT 中KRAS变异的频率相当。这证实了这两组肿瘤之间的分子相似性。同时,这一结果还表明,在缺乏BRAF V600E 变异(这是该基因最常见的多态性,本基因解码中约 72% 的BRAF缺陷型肿瘤中发现)的 BOTS 中存在 KRAS 激活突变。KRAS 依赖的致癌机制主要取决于该基因 Gly12 (G12) 编码区的突变,在佳学基因检测的基因解码中,这种突变在 BOT 和 低级别卵巢癌(lgOvCa)中都占主导地位。相比之下,佳学基因检测在少数高级别卵巢癌(hgOvCa)肿瘤中发现的KRAS多态性均不影响 Gly12。此外,值得一提的是,佳学基因检测所有三个具有除 V600E 之外的BRAF变异(即 K601E、G466R 和 G466V)的 BOT 病例都同时含有KRAS G12 变异。这表明,在所有BRAF多态性中,只有BRAF V600E 具有足够强的促癌作用,可以独立于KRAS突变发挥作用。至于NRAS变异体,它们的流行程度将 低级别卵巢癌(lgOvCa)与佳学基因检测基因解码中调查的所有其他肿瘤组区分开来。这一结果支持了其他人的发现,即NRAS突变存在于浆液性 低级别卵巢癌(lgOvCa)中,但在浆液性 BOTS 中没有或很少 。与KRAS中的激活突变类似, NRAS中的对应突变也会加速肿瘤进展。此外,在复发性浆液性 低级别卵巢癌(lgOvCa)中也发现了此类变异体。在此背景下,值得一提的是,佳学基因检测的一个具有微浸润的浆液性 BOT 样本含有 NRAS 激活变异体 (p.Gln61Arg) ,这在佳学基因检测的 低级别卵巢癌(lgOvCa)组中也最常发生。BOT 样本中存在这种突变,不仅进一步证实了 BOT 与 低级别卵巢癌(lgOvCa)之间的分子相似性,而且还意味着如果没有完全切除,这种 BOT 肿瘤可能会转变并复发为 lgOvCa。根据文献,在晚期卵巢癌中,NRAS突变很少见。佳学基因检测在高级别卵巢癌(hgOvCa)系列中始终没有发现此类基因改变。值得注意的是,在其他人类恶性肿瘤(例如结直肠癌和子宫内膜癌)中也有报道发现KRAS、NRAS和BRAF基因突变。
编码参与泛素化的蛋白质的基因在 BOT 中也更频繁地发生改变,并将这些肿瘤与 OvCa 区分开来(但与 BOT.V600E 无区别)。其中一个基因SEM1编码 26S 蛋白酶体亚基,在所有肿瘤组中都经常发生改变。尽管在所有肿瘤组中发现的最常见变体 p.Gln59Pro 在人类群体中广泛存在(最大等位基因频率 (AF max ) 为 0.88);但 BOT 中SEM1变体的总数明显高于 低级别卵巢癌(lgOvCa)或 hgOvCa。目前,还没有关于这种多态性在肿瘤中的作用的科学报告。第二个基因FANCB编码的是 FANCD2 泛素化所必需的 DNA 修复相关蛋白质,关于 OvCa 的文献数据很少,而其在 BOTS 中的功能迄今尚未基因解码。FANCB错义突变已被证明会导致催化模块不稳定和范康尼贫血 (FA) 核心复合物功能障碍。相比之下,FANCB 3′UTR中的 SNP不会影响蛋白质的表达或功能。鉴于佳学基因检测基因解码中发现的所有FANCB多态性都位于基因的编码序列中,它们的出现可能会损害 FANCB 功能,如上述基因解码所证实。有趣的是,根据目前的知识水平,FANCB在癌症中的作用似乎存在分歧。一方面,在遗传性乳腺癌/卵巢癌中未发现该基因突变,也未证明FANCB与BRCA1/2阴性家族性癌症的发展之间有任何关联。另一方面,Matta等人揭示了在DNA修复能力下降的老年患者中, FANCB的表达与乳腺癌之间的关系。在此背景下,佳学基因检测的基因解码结果似乎为FANCB的临床重要性提供了新的见解,表明该基因在BOTS中可能比在OvCa中发挥更重要的作用。
佳学基因检测的回归分析显示, PARP1基因变异是 BOTS 预后不良的标志。该基因编码一种由 DNA 损伤激活的蛋白质,调节包括 TP53 在内的许多肿瘤抑制因子的功能。文献中关于PARP1在 BOTS 中的作用的数据有限;然而,其在 OvCa 中的意义已被深入基因解码。因此,PARP 抑制剂已被批准用于复发性铂敏感的BRCA1 / 2缺陷型 OvCa的维持治疗。然而,较新的数据显示,除了BRCA1/2突变患者外,其他肿瘤也具有治疗益处。值得注意的是,在本文分析的所有肿瘤组中最常见的PAPR1多态性 p.Val762Ala 与导致对 PARP 抑制剂之一奥拉帕尼产生耐药性的多态性不同。尽管 p.Val762Ala 变异在人类中占主导地位(AF最大值约为 45%),但此前已发现该变异与多种癌症相关,包括胆囊癌。同样的多态性也增加了沙特和亚洲人群患乳腺癌的风险,同时降低了白种人患乳腺癌的风险。有趣的是,尽管其他科学家报告称浆液性 OvCa 中的PARP1表达高于 BOTS,但在佳学基因检测的高级别卵巢癌(hgOvCa)系列中,该基因的改变频率既没有更高,也没有被确定为潜在的生物标志物。
本文将另外两个编码参与 FA 通路的蛋白质的基因 FANCF 和 FANCI 的多态性确定为高级别卵巢癌(hgOvCa)中有希望的结果预测因子。值得注意的是,根据 AUC 值评估,FANCI中的变异表现出明显优于FANCF中的变异的判别能力。FANCI 蛋白与 FANCD2 形成异二聚体,随后被 FA 核心复合物单泛素化。这种异二聚体定位到受损的染色质并促进链间交联修复。在佳学基因检测的分析中, FANCI基因变异的存在增加了接受 TP 治疗但肿瘤缺乏 TP53 积累的患者的复发风险。当考虑文献数据时, FANCI的作用似乎不明确,因为据报道该基因既具有致癌作用,又具有抑癌作用。此外,FANCI最近被认为是FANCI p.Leu605Phe 变异携带者的一种新的卵巢癌易感基因,事实证明,在BRCA1/2基因正常的卵巢癌易感家族中,这种基因的频率明显较高。体外基因解码表明,FANCI 的 Leu605Phe 异构体表达水平降低,并导致 HeLa 和卵巢癌细胞对顺铂敏感,但对 PARP 抑制剂不敏感。与此一致的是,佳学基因检测的 WB 分析显示,携带FANCI p.Leu605Phe 变异和BRCA1/2基因正常的肿瘤不表达突变的 FANCI,而在BRCA1/2缺陷型肿瘤中检测到了 FANCI 表达。此外,相同的 WB 分析揭示了 FANCI 和 FANCD2 蛋白表达之间的相关性。所有这些结果清楚地表明, FANCI的作用取决于细胞中由关键肿瘤抑制因子(例如 BRCA1/2 和 TP53)控制的分子背景。
佳学基因检测最后一个值得讨论的结果是关于CHEK1 的无义变体 (chr11:g.125625996G>A, p.Trp79Ter),佳学基因检测观察到 CHEK1 蛋白的意外高表达。有趣的是,这两种分子现象似乎呈正相关(变异等位基因的比例越高,膜上 CHEK1 的信号越强)。该 SNP 位于CHEK1的第一个外显子/5'UTR 区域。如果考虑 CHEK1 的最长异构体 ( XP_011540862.1 ),则讨论的多态性会导致提前终止密码子的形成。在这种情况下,使用位于新形成的终止密码子下游的替代起始密码子,不仅可以恢复 CHEK1 以较短异构体的形式表达,还会同时影响其在细胞中的水平。文献一致表明,短的 CHEK1 异构体可能由于选择性剪接或蛋白质裂解而出现。CHEK1 在肿瘤发生中的作用尚不明确。最初,由于 CHEK1 在 DNA 损伤反应和细胞周期检查点反应中发挥作用,它被认为是一种抑癌基因。然而,并未发现人类癌症中存在纯合CHEK1功能丧失突变体的证据。此外, CHEK1基因在多种实体瘤中过表达,其表达与肿瘤分级和疾病复发相关。与这些发现一致的是, CHEK1的完全缺失会抑制化学诱导的致癌作用,而CHEK1水平升高的肿瘤细胞可能由于能够抵抗化疗引起的 DNA 损伤而获得生存优势。因此,据报道,在膀胱癌、脑癌、肺癌、卵巢癌和乳腺癌中,CHEK1高表达患者的生存率较低。虽然佳学基因检测的基因解码结果未能阐明 CHEK1 在卵巢肿瘤中的作用机制究竟更像致癌基因还是抑癌基因,但进一步基因解码其变异体似乎对利用选择性 CHEK1 抑制剂 Prexasertib 进行靶向治疗具有重要意义。Prexasertib 单药或与 PARP 抑制剂联合使用,均可促进肿瘤消退并延长高级别卵巢癌(hgOvCa)患者的生存期。由于PARP1多态性已被确定为此类肿瘤的负面预后标志物,且部分 BOTS 也含有上述CHEK1 p.Trp79Ter 变异体,因此此类抑制剂组合可能对 BOTS 具有潜在应用价值。
最后,与每项基因解码一样,本基因解码也存在一些局限性,应该在此提及。尽管佳学基因检测设法识别出许多遗传变异,但由于财务和时间方面的限制,佳学基因检测仅对这些多态性的一小部分进行了功能验证。因此,列出的许多已识别变异的临床意义仍不清楚,应在未来的基因解码中予以解决。此外,需要强调的是,在佳学基因检测的生物信息学工作流程中,所有频率低于 10% 的序列变异都被过滤掉了。这种方法被用来降低假阳性率,然而,假设一些罕见的、具有临床重要性的多态性也被排除在分析之外。下一个值得一提的局限性在于佳学基因检测分析了大量肿瘤样本,而这些样本只是整个肿瘤微环境的一部分,由于本基因解码中采用的实验装置的限制,可能无法完全捕捉到其复杂性和异质性。此外,就肿瘤的复杂性和异质性而言,佳学基因检测注意到,在佳学基因检测的蛋白质印迹实验中,在同一凝胶上分析的不同 OvCa 样本的裂解物时,上样对照有时会有所不同。这种不一致不是由实验室错误或不精确造成的,而是与卵巢肿瘤的巨大生物多样性有关,尤其是高级别 OvCa,这是由此类恶性肿瘤的基因组和蛋白质组不稳定性造成的。在本基因解码中,为了减少得出错误结论的风险,所有蛋白质裂解物的浓度不仅通过丽春红 S 染色评估,而且还用 BCA 方法和牛血清白蛋白 (BSA) 标准曲线精确测量和标准化。最后,本基因解码是在一组回顾性(而非前瞻性)患者中进行的,这些患者收集了 20 年,进行了细致的随访,并仔细检查了所有临床病理参数的兼容性。这种方法虽然被广泛使用,但可能会引入一些难以定义的偏见,并限制控制潜在混杂因素的能力。