
引言
CRISPR-Cas9系统由于其便于操作及靶向性等特点被用来开发为一系列基因编辑工具:比如核酸酶编辑(nuclease editing)、碱基编辑(base editing)和先导编辑(prime editing)等【1】。其中,碱基编辑可以在不引起双链DNA断裂的情况下有效地在目标位点实现单个碱基的替换,在单碱基突变疾病治疗中展现出了广阔的应用前景【2-3】。然而,由于SpCas9的体积较大(1368个氨基酸),基于SpCas9介导的碱基编辑器都超过了单个腺相关病毒(AAV)载体包装的大小(~4.7 kb),这限制了其在体内基因编辑的应用【4-5】。因此,缩小碱基编辑器的大小对其在在体疾病治疗的应用至关重要。
为了解决这一问题,研究者们开始寻求用小Cas蛋白代替Cas9进行精确的基因编辑。最近,一系列微型RNA引导的基因组编辑系统,如Cas12f, Cas12m, Cas12n, TnpB和IscB等被发现并被改造成基因组编辑工具【6-16】。但Cas12家族蛋白以及TnpB蛋白无法改造成为nickase变体来开发高效的微型碱基编辑器。2021年张峰团队发现了IS200/IS605转座子超家族中的一类RNA引导核酸酶IscB,IscB-ωRNA系统被认为是CRISPR-Cas9系统的祖先,IscB与Cas9具有相似的结构域,但大小只有SpCas9的1/3,满足了AAV作为基因治疗载体的包装能力【15】。其中,天然的OgeuIscB被报道在哺乳动物细胞内不到2%的编辑活性,需要进一步优化提高其编辑效率。2022年,可爱龙和张峰团队先后解析了IscB-ωRNA的结构及其切割DNA的机制,这为对IscB-ωRNA系统的优化提供了有利的基础【17,18】。
2024年7月8日,上海科技大学马涵慧课题组在学术期刊Nature Chemical Biology上发表了题为Engineering miniature IscB nickase for robust base editing with broad targeting range的研究论文,该研究通过工程化改造OgeuIscB-RNA系统并融合DNA双链结合蛋白Sso7d开发了高效的微型碱基编辑器-SIminiBEs。SIminiBEs显示了强大的C到T或A到G的碱基转换能力,并表现出广泛的靶向范围,近乎不受靶向位点限制(Near TAMless)。

该研究首先基于IscB-ωRNA-DNA复合体的结构,设计了26个突变体,经过多轮的筛选,得到了活性最高的突变体IscBnQM。同时,作者删去了不与IscB蛋白和DNA发生相互作用的ωRNA区域,得到了活性最高的突变体opti ωRNA。作者将基于IscBnQM和opti ωRNA的碱基编辑器命名为IminiBEs,并分别构建了IminiCBE, IminiABE, IminiCGBE以及IminiAYBE。其中,IminiCBE(平均编辑效率为67%)和IminiABE(平均编辑效率为52%)分别实现了高效的C到T或A到G的碱基编辑效率。而IminiCGBE和IminiAYBE中C到G或A到Y的平均编辑效率低于20%。为了解决在一些靶向位点编辑效率仍然较低的问题,作者将非特异性DNA结合蛋白Sso7d与IminiBEs融合后,进一步将低效位点的编辑效率提高约2 ~ 3倍,并称之为SIminiBEs。

图1:IminiBEs和SIminiBEs示意图(Credit: Nature Chemical Biology)
此外,通过TAM(靶邻近基序)识别实验发现,相较于野生型IscB nickase的典型NWRRNA(N代表任何碱基,W代表碱基A/T, R代表碱基A/G)TAM位点,IminiCBE和SIminiCBE对+2或+6位点没有了明显的偏好,而对+3和+4位点RR的偏好也明显减弱,近乎不受靶向位点限制(Near TAMless),极大地拓宽了IminiCBE和SIminiCBE在基因组中的靶向范围及其应用场景。
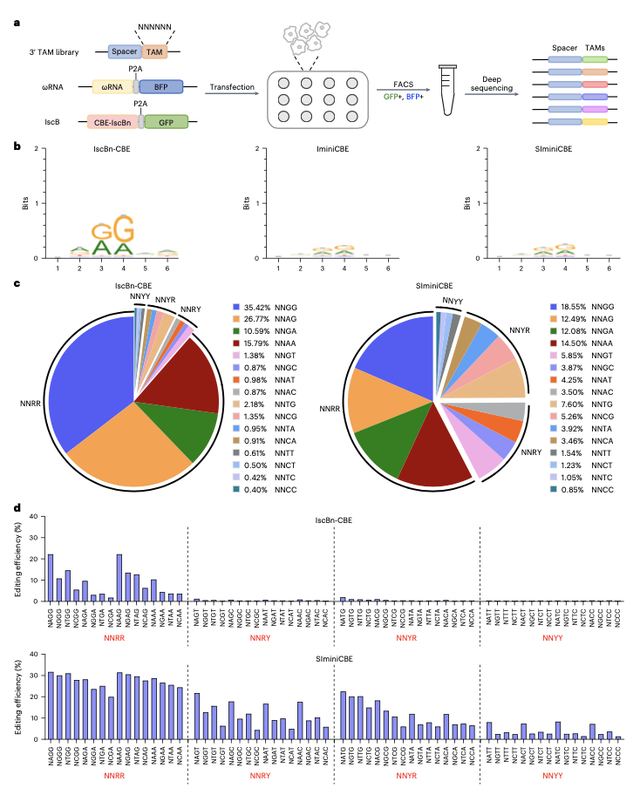
图2:IscBn-CBE, IminiCBE和SIminiCBE的TAM分析(Credit: Nature Chemical Biology)
总体而言,作者开发的IminiBEs和SIminiBEs是高效、宽靶向性的微型碱基编辑器,扩展了我们用于基因组编辑的微型CRISPR工具箱,有助于解决目前由于碱基编辑器太大而无法被单个AAV载体递送的难题。
参考文献
Anzalone, A. V., Koblan, L. W. & Liu, D. R. Genome editing with CRISPR–Cas nucleases, base editors, transposases and prime editors. Nature Biotechnology 38, 824–844 (2020).Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A. & Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420–424 (2016).Landrum, M. J. et al. ClinVar: public archive of interpretations of clinically relevant variants. Nucleic Acids Research 44, D862–D868 (2015).Dong, J. Y., Fan, P. D. & Frizzell, R. A. Quantitative Analysis of the Packaging Capacity of Recombinant Adeno-Associated Virus. Human Gene Therapy 7, 2101–2112 (1996).Wu, Z., Yang, H. & Colosi, P. Effect of Genome Size on AAV Vector Packaging. Molecular Therapy 18, 80–86 (2010).Kim, D. Y. et al. Efficient CRISPR editing with a hypercompact Cas12f1 and engineered guide RNAs delivered by adeno-associated virus. Nature Biotechnology 40, 94–102 (2021).Xu, X. et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Molecular Cell 81, 4333-4345.e4 (2021).Wu, Z. et al. Programmed genome editing by a miniature CRISPR-Cas12f nuclease. Nature Chemical Biology 17, 1132–1138 (2021).Kim, D. Y. et al. Hypercompact adenine base editors based on a Cas12f variant guided by engineered RNA. Nature Chemical Biology 18, 1005–1013 (2022).Zhang, S. et al. TadA reprogramming to generate potent miniature base editors with high precision. Nature Communications 14, 413 (2023).Wu, T. et al. An engineered hypercompact CRISPR-Cas12f system with boosted gene-editing activity. Nature Chemical Biology (2023).Wu, W. Y. et al. The miniature CRISPR-Cas12m effector binds DNA to block transcription. Molecular Cell 82, 4487-4502.e7 (2022).Chen, W. et al. Cas12n nucleases, early evolutionary intermediates of type V CRISPR, comprise a distinct family of miniature genome editors. Molecular Cell 83, 2768-2780.e6 (2023).Karvelis, T. et al. Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease. Nature 599, 692–696 (2021).Altae-Tran, H. et al. The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases. Science 374, 57–65 (2021).Han, D. et al. Development of miniature base editors using engineered IscB nickase. Nature Methods 20, 1029–1036 (2023).Schuler, G., Hu, C. & Ke, A. Structural basis for RNA-guided DNA cleavage by IscB-ωRNA and mechanistic comparison with Cas9. Science 376, 1476–1481 (2022).Kato, K. et al. Structure of the IscB–ωRNA ribonucleoprotein complex, the likely ancestor of CRISPR-Cas9. Nature Communications 13, 6719 (2022).https://www.nature.com/articles/s41589-024-01670-w责编|探索君
排版|探索君
文章来源|“BioArt”
End
