
引言
空间转录组技术的进展为研究肿瘤微环境中的细胞互作提供了重要见解。该技术产生的数据不仅包含组织的空间基因表达,也涵盖了病理切片信息(H&E image)。目前,许多分析工具都在探索这些数据,主要用于细胞解卷(cell deconvolution)和聚类(clustering)。然而,现有的工具大多只考虑基因表达信息,却忽略了图像中的细胞形态特征。特别是在mRNA捕获率不一的区域,仅依赖基因表达分析难以准确识别不同的区域和细胞类型,从而影响分析的准确性。因此,结合病理切片上的细胞形态学特征与空间基因表达数据,可以更精确地识别肿瘤微环境中的不同区域和细胞类型。
2024年8月25日,埃默里大学(Emory University)的胡健团队与得克萨斯大学MD Anderson肿瘤医院的王凌华团队在Nature Communications上发表论文METI: Deep profiling of tumor ecosystems by integrating cell morphology and spatial transcriptomics,介绍了一种新的空间转录组数据分析框架。胡健和王凌华担任共同通讯作者,博士生蒋嘉汇是该论文的第一作者。
 METI是一种机器学习方法,它通过整合空间基因表达数据和病理学组织学特征,系统性地分析肿瘤生态系统。该方法结合了从先验知识中获得的基因表达和细胞形态学特征,能够准确识别并标注肿瘤细胞、免疫细胞及其亚群。如图1所示,METI以空间转录组数据作为输入,包括基因的空间表达矩阵、对应的H&E染色组织切片图像,以及每个点位的X,Y坐标。它结合图像分割和基因表达的方法识别细胞类型,并整合两者结果以减少单一方法的误差。METI的分析框架包括四个递进的模块:模块1能识别正常和癌前细胞,如胃中的杯状细胞【1-3】;模块2负责识别肿瘤细胞富集区域,并表征其细胞状态的异质性;模块3聚焦于T细胞,包括CD4+ 和CD8+ T细胞及其不同状态,如调节性T细胞(Treg)和耗竭性T细胞(Tex);模块4专注于癌症相关成纤维细胞(CAFs),并细分其亚型,包括myCAFs、iCAFs和apCAFs【4-8】。这些细胞在癌症进展和治疗耐受中起重要作用【9-11】。对每个模块,METI都能生成特定细胞类型的图像分割结果、基因表达图、整体分布,以及细胞密度的3D空间可视化。
METI是一种机器学习方法,它通过整合空间基因表达数据和病理学组织学特征,系统性地分析肿瘤生态系统。该方法结合了从先验知识中获得的基因表达和细胞形态学特征,能够准确识别并标注肿瘤细胞、免疫细胞及其亚群。如图1所示,METI以空间转录组数据作为输入,包括基因的空间表达矩阵、对应的H&E染色组织切片图像,以及每个点位的X,Y坐标。它结合图像分割和基因表达的方法识别细胞类型,并整合两者结果以减少单一方法的误差。METI的分析框架包括四个递进的模块:模块1能识别正常和癌前细胞,如胃中的杯状细胞【1-3】;模块2负责识别肿瘤细胞富集区域,并表征其细胞状态的异质性;模块3聚焦于T细胞,包括CD4+ 和CD8+ T细胞及其不同状态,如调节性T细胞(Treg)和耗竭性T细胞(Tex);模块4专注于癌症相关成纤维细胞(CAFs),并细分其亚型,包括myCAFs、iCAFs和apCAFs【4-8】。这些细胞在癌症进展和治疗耐受中起重要作用【9-11】。对每个模块,METI都能生成特定细胞类型的图像分割结果、基因表达图、整体分布,以及细胞密度的3D空间可视化。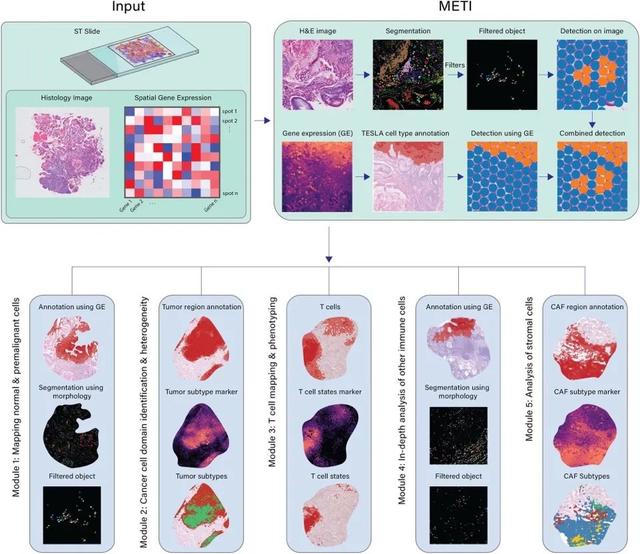 图1:METI算法功能介绍(Credit: Nature Communications)METI利用图像形态学和基因表达的结合优势在定位杯状细胞的案例中得以展示。图2a显示了病理医生对杯状细胞的注释,这些细胞主要富集在四个区域。通过基因表达分析,METI成功识别了与标注相符的杯状细胞高表达区域1和2(图2b和2c)。然而,区域3和4由于mRNA总体数量较低,未能通过基因表达直接识别(图2d)。METI采用图像分割技术,并根据文献报道的杯状细胞的形态特征设置了多个过滤条件,如细胞面积和长宽比,以精准区分并保留这些细胞。图2e展示了经过过滤后的杯状细胞。METI将这些分割结果转换为细胞富集注释(图2f),并将其与基因表达结果相结合(图2g),最终成功捕捉到了区域4的杯状细胞。这一成果展示了METI在综合图像和分子数据分析方面的高效能力。综上所述, METI是一个整合图像形态和基因表达从而寻找细胞类型区域的工具。随着空间转录组在生物医学研究中的日益普及,我们希望METI能对研究人员的大规模空间转录组数据分析提供帮助。AI in Genomics lab 正在招收机器学习和基因组学的方向的博士生和博士后,详情请见https://jianhu-lab.org/。
图1:METI算法功能介绍(Credit: Nature Communications)METI利用图像形态学和基因表达的结合优势在定位杯状细胞的案例中得以展示。图2a显示了病理医生对杯状细胞的注释,这些细胞主要富集在四个区域。通过基因表达分析,METI成功识别了与标注相符的杯状细胞高表达区域1和2(图2b和2c)。然而,区域3和4由于mRNA总体数量较低,未能通过基因表达直接识别(图2d)。METI采用图像分割技术,并根据文献报道的杯状细胞的形态特征设置了多个过滤条件,如细胞面积和长宽比,以精准区分并保留这些细胞。图2e展示了经过过滤后的杯状细胞。METI将这些分割结果转换为细胞富集注释(图2f),并将其与基因表达结果相结合(图2g),最终成功捕捉到了区域4的杯状细胞。这一成果展示了METI在综合图像和分子数据分析方面的高效能力。综上所述, METI是一个整合图像形态和基因表达从而寻找细胞类型区域的工具。随着空间转录组在生物医学研究中的日益普及,我们希望METI能对研究人员的大规模空间转录组数据分析提供帮助。AI in Genomics lab 正在招收机器学习和基因组学的方向的博士生和博士后,详情请见https://jianhu-lab.org/。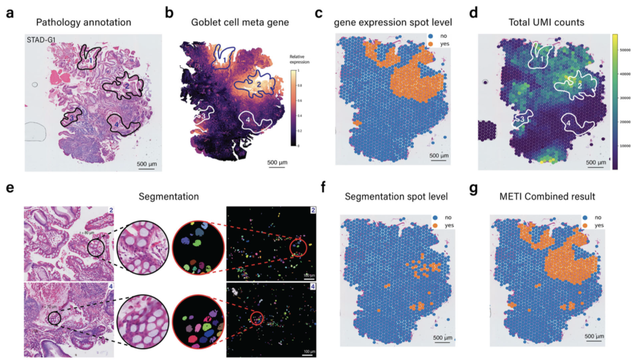
图2:结合图像形态学和基因表达定位杯状细胞(Credit: Nature Communications)
软件链接:https://github.com/Flashiness/METI参考文献
[1] Correa, P., M.B. Piazuelo, and K.T. Wilson, Pathology of gastric intestinal metaplasia: clinical implications. The American journal of gastroenterology, 2010. 105(3): p. 493.[2] Leung, W. and J. Sung, Intestinal metaplasia and gastric carcinogenesis. Alimentary pharmacology & therapeutics, 2002. 16(7): p. 1209-1216.[3] Jencks, D.S., et al., Overview of Current Concepts in Gastric Intestinal Metaplasia and Gastric Cancer. Gastroenterol Hepatol (N Y), 2018. 14(2): p. 92-101.[4] Ohlund, D., et al., Distinct populations of inflammatory fibroblasts and myofibroblasts in pancreatic cancer. J Exp Med, 2017. 214(3): p. 579-596.[5] Elyada, E., et al., Cross-Species Single-Cell Analysis of Pancreatic Ductal Adenocarcinoma Reveals Antigen-Presenting Cancer-Associated Fibroblasts. Cancer Discov, 2019. 9(8): p. 1102-1123.[6] Hu, B., et al., Subpopulations of cancer-associated fibroblasts link the prognosis and metabolic features of pancreatic ductal adenocarcinoma. Ann Transl Med, 2022. 10(5): p. 262.[7] Sahai, E., et al., A framework for advancing our understanding of cancer-associated fibroblasts. Nature Reviews Cancer, 2020. 20(3): p. 174-186.[8] Geng, X., et al., Cancer-Associated Fibroblast (CAF) Heterogeneity and Targeting Therapy of CAFs in Pancreatic Cancer. Front Cell Dev Biol, 2021. 9: p. 655152.[9] Saw, P.E., J. Chen, and E. Song, Targeting CAFs to overcome anticancer therapeutic resistance. Trends Cancer, 2022. 8(7): p. 527-555.[10] Asif, P.J., et al., The role of cancer-associated fibroblasts in cancer invasion and metastasis. Cancers, 2021. 13(18): p. 4720.[11] Feng, B., et al., Cancer-associated fibroblasts and resistance to anticancer therapies: Status, mechanisms, and countermeasures. Cancer Cell International, 2022. 22(1): p. 166.https://www.nature.com/articles/s41467-024-51708-9责编|探索君
排版|探索君
文章来源|“BioArt”
End
